Wolf-Ekkehard Lönnig:
EVOLUTION DURCH GEN-DUPLIKATIONEN?
The mechanism of gene duplication as the means to acquire new genes with previously nonexistent functions is inherently self limiting in that the function possessed by a new protein, in reality, is but mere variation of the preexisted theme. ...A new protein by this mechanism invariably retains substantial amino acid sequence homology and, therefore, functional relatedness with the immediate ancestor.
Susumu Ohno
At all events, tandem duplication does not
solve the evolutionary
dilemma. It might give a rapid increase in the quantity of genetic
material, but it only
does so by being highly repetitive, and this will not give a sequence of
'quantum jumps' in the forms of plants and animals, such as is needed to
provide for the divergent evolutionary branches...
Hoyle and Wickramasinghe Da die Neuentstehung von Genen durch
Genduplikation mit nachfolgenden
Mutationen des Reserve-Gens nichts anderes ist als eine Variante der
mutativen Abänderungg von Genen generell, liegen auch die
Haupteinwände dagegen auf der gleichen
Ebene...
Ferdinand Schmidt
Der den Vorbemerkungen folgende Text ist aus meinem Buch ARTBEGRIFF, EVOLUTION UND SCHÖPFUNG, 3. korrigierte Auflage 1993, Kapitel V.2. Gen-Duplikationen, pp. 420-437 und 558-566, entnommen und überarbeitet worden; vgl. im Zusammenhang mit diesen Ausführungen den Beitrag Mutationen: Das Gesetz der rekurrenten Variation).
VORBEMERKUNGEN UND EINIGE HAUPTPUNKTE (2001)
Erstens: Gen-Duplikationen sind nur ein Spezialfall des Gesetzes der rekurrenten Variation. Auch Gen-Duplikationen und -Amplifikationen treten in Populationen der verschiedensten Arten regelmäßig auf, teilweise sogar mit höheren Frequenzen als andere Mutationen. Nach Roberts und Broderick beträgt die Gen-Duplikationsrate bei Drosophila 2 x 10-5 ("as the probability...of a genome carrying a newly generated tandem sequence at a specific locus. The frequency...is some twenty times the average mutation rate for a specific locus"). Durch mutagene Agenzien wird diese Rate weiter erhöht. - In tierischen und menschlichen Gewebekulturen können bis zu 10% aller Zellen eine Duplikation aufweisen ("Amplification occurs at a frequency greater than the spontaneous point mutation rate..." - B. Lewin, 1994, p. 1087).
Mit anderen Worten sind in den
umfangreichen Mutagenese-Experimenten mit Drosophila und in der
Züchtungsforschung über die Gen- (und sonstigen) Mutationen
hinaus auch unzählige Gen-Duplikationen und Gen-Amplifikationen
induziert worden. In dem negativen Gesamtresultat der
Mutationszüchtung und der experimentellen Artbildungsfrage sind also
die Gen-Duplikationen umd -Amplifikationen bereits voll enthalten. Nach neueren Ansätzen ist die
Duplikationsrate
ebenfalls viel
höher als von den Autoren erwartet und "the vast majority of gene duplicates are silenced within a few
million
years". Der Rest könnte dann zur Bildung von Isolationsmechanismen beitragen. [Vgl. Lynch und Conery
(2000) in Science 290, pp. 1151-1155 und Pennisis Kommentar im selben Heft dazu; siehe weiter die Diskussionen
von Long und Thornton, sowie Zhang et al. versus Lynch and Conery in Science 293, pp. 1551 ff. (31. August
2001)]. Statt jedoch empirische Studien und Beobachtungsergebnisse als Grundlage für die tatsächlichen
Genduplikationsraten zu
nehmen und diese Resultate auf die Genduplikationsfrage bei ihrer Interpretation ganzer Genome anzuwenden, gehen
die
neuen Ansätze in mehreren Punkten von fraglichen theoretischen Voraussetzungen aus und
kommen dabei - im Gegensatz zu den oben zitierten Beobachtungen - zu niedrigeren Duplikationsraten ("duplications
occur as often as single-base changes within genes..." - Pennisi).
Klyce (2000) kommentiert den evolutionstheoretischen Hauptpunkt der Arbeit von Lynch und Conery
wie folgt:
"Since 1970, when Susumo Ohno published Evolution by Gene Duplication, this mechanism has
become part of Darwinian orthodoxy. If gene duplication can lead to speciation, as the study concludes, it would
indeed promote genetic variation. But genetic variation is not the same as macroevolutionary progress, which
requires lengthy new genetic programs. If gene duplication can produce them, one would expect a study like this
one to detect evolutionary pathways connecting genes with quite different functions. Yet there is no mention of
any evidence for such pathways. The only evolutionary pathways confirmed are between members of pairs with closely
related functions. We think the ability of Darwinian evolution to produce sustained macroevolutionary progress in
a genetically closed system still rests on weak support" (Brig Klyce, Panspermia Nov./Dec. 2000).
Zweitens: In noch größerem
Ausmaß sind Gen-Duplikationen und -Amplifikationen in pflanzlichen
Zell- und Gewebekulturen erzeugt worden. Das hatte den Vorteil, dass aus
diesen
Kulturen direkt auf brauchbare Eigenschaften selektierte Pflanzen
regeneriert werden konnten. Diese umfangreichen Experimente haben jedoch,
selbst
unter
Einbeziehung der
Fusion artverschiedener Protoplasten, so
selten zu irgendwelchen brauchbaren Ergebnissen geführt, dass auch
diese - nach evolutionstheoretischen Voraussetzungen - fest erhoffte
Revolution in der Pflanzenzüchtung ausgeblieben
ist (vgl. unten: "The Revolution that Failed"). Es bedarf kaum einer
Erwähnung, dass auch in diesen
Versuchen mit Milliarden von Genduplikationen weder primäre noch
sekundäre neue Pflanzenspezies
entstanden sind. Während Sie diese Zeilen studieren,
lieber Leser, werden zusätzlich rund um den Globus von Tausenden von
molekularbiologischen Arbeitsgruppen bei der Transformation und
Regeneration von Kulturpflanzen aus Zellkulturen weitere Milliarden
und Abermilliarden von Gen-Duplikationen induziert. Jedoch haben
auch die biologisch versiertesten Gentechnologie-Gegner, die nur alle
denkbaren
Möglichkeiten in die Diskussion einer eventuellen Bedrohung des
ökologischen Gleichgewichts durch transformierte Pflanzen
eingebracht haben, noch nie die dabei massenhaft entstehenden
Gen-Duplikationen
für eine Gefahr gehalten - etwa als eine gigantische Beschleunigung
der Evolution durch den Menschen mit verheerenden Auswirkungen auf die
Biozönosen dieser Erde. Vielmehr konzentriert sich die Kritik
schwerpunktmäßig auf die möglichen biologischen Folgen des
Transfers von hochspezifischen DNA-Sequenzen (Genen) von einer
Art auf
eine andere. Denn es gibt kein einziges Beispiel, dass solche neuen
hochspezifisch-funktionalen DNA-Sequenzen bei den großen Zahlen
von Gen-Duplikationen der aus Zellkulturen erhaltenen Pflanzen
entstehen. Drittens: Während Pflanzen einen relativ
großen Toleranzbereich bei Duplikationen aufweisen können, sind
die Grenzen bei Säugetieren enger gesteckt. - Bei Pflanzen sind
Duplikationen und Amplifikationen vor allem für den Teil des
rekurrenten Variationsspektrums verantwortlich, der nach mehreren
großen Mutagenese-Experimenten das Variationsspektrum nach 99%iger
Sättigung quantitativ weiter asymptotisch gegen Null laufen
lässt. Viertens: Dass Duplikationen mit ausgesprochen
negativen Auswirkungen auf die betroffenen Organismen verbunden sein
können, das zeigen besonders eindringlich die Ploidiemutationen und
Trisomien des Menschen: "Menschliche Embryonen mit einem
dreifachen Chromosomensatz (Triploidie) sterben meist schon in
einer frühen Phase ihrer Entwicklung. Selbst das dreifache Vorkommen
eines einzelnen Chromosoms (Trisomie) verursacht beim Menschen
meist den Tod des betroffenen Embryos in den ersten Monaten der
Entwicklung. Nur wenige Embryonen mit einer Trisomie überleben bis
zum Ende einer Schwangerschaft. Dazu gehören die Trisomien der
Chromosomen 13, 18 und 21. Doch auch hier leiden die betroffenen
Neugeborenen unter schweren Entwicklungsanomalien, die unter anderem eine
geringere Lebenserwartung zur Folge haben" (R. Knippers, Molekulare
Genetik
1997, p. 155). Das trifft weiter auch auf die
Verdoppelung von Chromosomenstücken zu:
"Like some deletions, duplications of some
human chromosomes cause syndromes of phenotypic abnormalities. A person
afflicted with a duplication syndrome has three copies of the duplicated
region, whereas other chromosome regions are present in two copies as
usual. Humans homozygous for duplications are unknown in medical
genetics" (Griffith et al. 1993, p. 217). Auf der Genebene führen
Amplifikationen in
Proto-Onkogenen beim Menschen zu verschiedenen Krebsarten
("Gene amplification is frequently observed in association with human and
other animal cancers"- S.L.Wolfe 1993, p. 938; vgl. weiter die
einschlägigen Lehrbücher). Fünftens: Im Rahmen der Diskussion des
Themas Transposons, Evolution und Gen-Duplikationen haben wir 1997 die
grundlegende Frage
gestellt, ob wir überhaupt weitere redundante DNA-Sequenzen für
die Bildung neuer Genfunktionen brauchen:
"From the view of the selfish DNA
hypothesis one may ask, for instance, whether gene duplications are
necessary at all for the origin of new gene families. There seems to be
already so much 'junk' in the genomes of most plant and animal species by
the propagation of the many different transposable element families
themselves (including their inactive derivatives) as well as other selfish
DNA sequences that further duplications may appear more or less
unnecessary. More generally put, hardly any organism appears to be
in need of further redundant DNA" (Kunze, Saedler, Lönnig,
1997, p. 425 - Hervorhebung im Schriftbild nachträglich). Die Gen-Duplikationshypothese als
Ausgangspunkt für die Entwicklung neuer Sequenzen stammt aus einer
Zeit als zur Frage nach 'junk'-DNA und überhaupt nach
redundanten Sequenzen praktisch noch nichts bekannt war. So
rechnete(n) bei in etwa gleichbleibeneden Schätzungen der
Gesamt-DNA-Menge Ernst Mayr und viele andere Forscher in der Mitte der
70er Jahre des
vorigen Jahrhunderts noch mit etwa 5 Millionen Genloci beim Menschen,
heute variieren nach den neuesten Daten die Schätzungen zwischen nur
30 000 und
40 000 (und nach Hazeltine etwa 120 000) Genen.
Sechstens: Die Homöobox-Gene sind in den
letzten Jahren sowohl in der Wissenschaft als auch in der
Öffentlichkeit
als Bestätigung der Evolutiontheorie durch Gen-Duplikationen viel
beachtet worden. Worum handelt es sich? Obwohl sich bei Drosophila die von den
Homöobox-Genen
kodierten Proteine stark
in ihrer Aminosäure-Sequenz und -Größe
voneinander unterscheiden,
weisen sie doch einen gemeinsamen aus 60 Aminosäureresten bestehenden
äußerst ähnlichen Abschnitt
auf: die Homöobox. Da nun diese Homöobox in allen Genen der
entsprechend benannten Genfamilie auftritt, liegt eine Erklärung
durch
zufällige Domänen- und Gen-Duplikationen nach
neodarwinistischen Voraussetzungen
nahe. Darüber hinaus ist auch häufig der Eindruck entstanden,
als
könne man allein mit der Vervielfätigung von Homöoboxgenen
ganz neue Baupläne erzeugen. Zunächst: Welche Funktion haben
Homöoboxgene? "Homeoboxgene haben sich als genetische
Schalter erwiesen. Sie werden auch als "Mastergene" bezeichnet, deren
Genprodukte in einem bestimmten Bereich des Embryos die Bildung einer
komplexen Struktur (etwa eines Körperteils) an- oder abschalten. Die
Schalter für vergleichbare Entwicklungsprogramme können dabei in
verschiedenen Grundtypen ähnlich sein. Das...Master-Kontrollgen
eyeless
ist in Maus, Mensch und Fruchtfliege bis in die Exon-Intron-Struktur
hinein erstaunlich ähnlich. Die tiefgreifenden
Sequenzähnlichkeiten der homologen Homeoboxgene gaben Anlaß zu
der Spekulation, daß die Enstehung der Extremitäten oder der
vielen unterschiedlichen Augentypen in Insekten, Wirbeltieren oder
Tintenfischen jeweils auf einen Ursprung zurückgehen. Makroevolution umfaßt die Entstehung
neuartiger
Strukturen. Das bloße An- oder Abschalten vorhandener
Strukturen in
anderen Zusammenhängen hilft bei dieser Frage nicht viel weiter. Bei
Maus und Fruchtfliege ist eyeless für das Anschalten des
Programms
"Augen-Bau" verantwortlich. Die Ähnlichkeit der beiden Gene geht
soweit, daß das entssprechende Mausgen, wenn es durch
molekularbiologische Methoden im Fruchtfliegenembryo exprimiert wird, dort
ein Fruchtfliegenauge (und kein "Mausauge") induziert. Das
bedeutet: Homeoboxgene an sich haben nichts mit dem Bauplan einer
komplexen Struktur zu tun" (Junker and Scherer 1998,
p. 124; Kursiv von den Verfassern). Oder für viele Fälle noch etwas genauer
gefasst: "Homeoboxgene
sind nur einer kleiner Teil des Bauplans einer komplexen Struktur" (Junker und
Scherer 2001, p. 125).
Nun weisen zum Beispiel Säugetiere
mit 39 für die Verwirklichung des Grundbauplans notwendigen
Homöoboxgenen - (dort
als
Hox-Gene bezeichnet, die
über die Hox-Domäne hinaus ähnliche
(Gesamt-)Sequenzen in zwei- bis
vierfacher Ausgabe zeigen) - sowie mit mehr als 300 weiteren
Homöoboxgenen offensichtlich eine wesentlich höhere Anzahl von
Angehörigen dieser
Genfamilie auf als Insekten und
andere
Lebensformen (Drosophila exprimiert zunächst 8
Homöoboxgene in der Embryonalentwicklung und ca. 90
weitere Glieder der Genfamilie sind bei der Fliege festgestellt
worden): Aufgrund
der Homöobox und der zahlenmäßigen Unterschiede zwischen
verschiedenen Lebensformen etc. sind die oben erwähnten
starken evolutionstheoretischen Behauptungen zu Duplikationen von
Homöoboxgenen geäußert worden.
Wie
gerade
ausgeführt,
haben Homöoboxgene jedoch hauptsächlich Schalterfunktionen. -
Zur
Veranschaulichung der Problematik ein Beispiel: Wer glaubt, allein mit der
Verfielfältigung von Schaltern, die Heizungsanlage eines
Wohnhauses in ein Kohle-
oder Kernkraftwerk umwandeln zu können, hat die wichtigsten Fragen in
diesem
Zusammenhang offenbar übersehen. In den genetischen Kaskaden und
Netzwerken sind Homöoboxgene oft Tausenden von weiteren spezifischen
Genfunktionen vorgeschaltet (beim Insektenauge etwa 2000, beim Menschen
belaufen sich die Schätzungen auf etwa 6000 weitere Gene). Mit der
Amplifikation von Homöoboxgenen allein schafft man keine neuen
Baupläne. Bei dieser Frage ist weiter zu bedenken, dass
auch die Homöoboxgene von Drosophila und anderen Organismen im
Zuge der regelmäßig auftretenden Gen-Duplikationen und
-Amplifikationen immer wieder vervielfältigt werden - offenbar mit
Funktions- und Selektionsnachteilen. Andernfalls gäbe es bei den
unterschiedlichen Arten keine festen Zahlen der Homöoboxgene.
Siebentens: Vergleicht man nun die Anzahl
und
Struktur der
Homöobox- und zahlreicher weiterer Gene verschiedener
Organismen, so
stellt man
selbstverständlich "Duplikationen" bei sämtlichen
Genfamilien fest (genauer sollte man
vielleicht von Gen- oder Sequenz-Duplikaten mit
unterschiedlich abgeänderten
Nukleotidfolgen
sprechen), - diese "Duplikationen" sind jedoch in ihren funktional
notwendigen Bereichen ebensowenig
Zufalls-Duplikationen
wie die im vorliegenden Satz sechsmal auftretende Buchstaben-Sequenz
"Duplikat" auf
Zufalls-Duplikationen beruht (Details siehe unten; vgl. zum
evolutionstheoretisch schwierigen Phänomen
der Konstanz
vieler Sequenzen auch die Diskussion zum Thema:
Unerwartete
"Conservation" auf molekularer Ebene... sowie zur Homologiefrage
überhaupt, den
Schlussteil
der Diskussion zur
Vogelfeder: Kuhn,
ReMine
und Troll).
Achtens: Für den Umbau einer durch
Genverdoppelung erzeugten redundanten Sequenz (d.h. einer Sequenz, die
nicht mehr funktionieren muss) in eine völlig neue
spezifisch-funktionale Nukleotidfolge als Entstehungsmodus etwa der
mehr
als 5000
nicht voneinander ableitbaren Genfamilien, und zwar ohne Selektionsdruck
("While being ignored by natural selection, they are free to accumulate
random base substitutions, deletions and insertions" - Ohno), gelten die
bekannten Unwahrscheinlichkeiten (vgl. Artbegriff p. 359): "Zur Veranschaulichung möchten wir ein Beispiel
von J. F. Coppedge zitieren. Wie groß ist die Wahrscheinlichkeit,
dass jemand zufällig aus einer Box mit Unmengen von Einzelbuchstaben
unseres Alphabets nur einmal die Sequenz "the theory of
evolution" zieht? All that is needed is simply to get those
twenty-three letters and spaces in proper order, selecting them at random
from a set of twenty-seven objects (twenty-six letters and one space). By
the multipication rule we learned, it will be 27 x 27 x 27...x 27 using
the
figure twenty-three times.
The probability when computed is 1 in approximately
834,390,000,000,000,000,000,000,000,000,000; that is one success in over 8
hundred million trillion trillion draws.
To get an idea of that number, let us imagine that chance is employing an
imaginary machine which will draw, record and replace the letters at the
speed of light, a BILLION draws PER SECOND! Working at that unbelievable
rate, chance could spell "the theory of evolution" once in something over
26,000,000,000,000,000 years on the average! ...a child could do it
in a few minutes. Chance would take more than five
million times as long as the earth has existed (if we use the
five-billion-year rounded figure which some evolutionists now estimate as
the age of the earth). Auf die genetische Ebene übertragen
heißt das für eine proteinkodierende Sequenz mittlerer
Länge (Coppedge, p. 159): With four kinds of nucleotides, and a
chain 1,200 long, the total of possible arrangements would be 41200,
which is approximately 10722. The
letters of a gene, however, are read in triplet codons (comprising
sixty-four kinds of triplets) of which there are 400 in this size
chain. If computed in this way, there would be a total of
64400possible
orders, and this turns out to the same as when figured by individual
letters, namely 10722.
Auch wenn wir einen Toleranzspielraum von
bis zu 50% einräumen, bleiben die Wahrscheinlichkeiten unvorstellbar
gering. S. Pullen hat speziell zur Unwahrscheinlichkeit der
Gesamtevolution durch Gen-Duplikationen umfangreiche Modellrechnungen
vorgelegt
(1998/2001:
Evolution
of New Genes from non-Random DNA - mit darauf folgenden 17
Internet-Unterkapiteln). Wenn auch diese Modelle sicher noch nicht das
letzte Wort zu diesem Thema
sind - die Situation ist in der biologischen Realität noch
wesentlich komplexer, da das gesamte Gebiet der spezifischen
raumzeitlichen Genregulation noch nicht in den Modellrechnungen
mitberücksichtigt worden ist - so ist doch der Trend der
Überlegungen
sowohl bei Coppedge als auch bei Pullen in voller Übereinstimmung mit
den beschreibenden und experimentellen
Ergebnissen der Mutationsforschung mit ihren insgesamt
in die Milliarden und Billionen gehenden spontanen und induzierten
Mutationen.
Zur
Widerlegung
von prinzipiellen Einwänden
gegen solche Wahrscheinlichkeitsansätze vgl. die Arbeiten des
Mathematikers William
A. Dembski, W. A. (1998a): The Design Inference: Eliminating Chance
Through Small Probabilities. Cambridge: Cambridge University
Press. Dembski , W. A. (1998b): Redesigning Science. In: Mere Creation,
pp. 93-112. Ed. W. A. Dembski. Downers Grove, Illinois: InterVarsity
Press. Im
übrigen
hat Walter
James ReMine in seinem Buch
THE BIOTIC MESSAGE (1993, p. 74ff.)
recht
gut begründet, warum er die grundsätzliche Ablehnung der
Wahrscheinlichkeitsfrage für die Evolution als einen Angriff auf
die
Wissenschaft selbst betrachtet (oder mit seinen Worten: "Some
evolutionists directly assault
probability science to brush it aside as insignificant or unreliable. This
is an assault on science itself" - siehe die daran anschließende
Diskussion). - Das ist bekanntlich nicht das erste Mal, dass Darwinisten
im Namen der Wissenschaft in Wirklichkeit einen Angriff auf diese
vornehmen; vgl. den Fall
Mendel,
insbesondere auch "Die
Reaktion des Darwinismus auf die 'Wiederentdeckung' der Mendelschen
Regeln". Damit wollen wir detailliert zur biologischen Frage
EVOLUTION DURCH GEN-DUPLIKATIONEN? übergehen, indem wir uns nun dem
überarbeiteten Text zum Thema Gen-Duplikationen aus der Arbeit
Artbegriff, Evolution und Schöpfung(1993) zuwenden: GEN-DUPLIKATIONEN Der Ansatz, dass bei der postulierten Evolution
Gen-Duplikationen eine besondere Rolle spielen würden, ist nicht
neu. Goldschmidt hatte sich schon vor Jahren mit dieser Frage im Anschluss an die Überlegungen Bridges (1918) auseinandergesetzt. Letzterer entwickelte die These, "dass ein verdoppeltes Gen unmittelbar neben dem anderen durch die Verdoppelung entstandenen identischen Gen läge, später aber durch Translokation an irgendeinen anderen Ort gelange und sich im Laufe der Zeit zu einem völlig anderen Gen umbilden könne. Das Vorhandensein des alten Gens lasse eine derartige Entwicklung eines neuen Gens zu, ohne dass dadurch eine Schädigung des Organismus einträte" (Goldschmidt 1955/1961, p.482). Auch sind in diesem Zusammenhang die Namen Lewis und Weir zu nennen, deren Überlegungen schon sehr stark an die heutigen molekularbiologischen Vorstellungen (einschließlich neutraler Gene - Weir 1953) erinnern, wie sie in neuerer Zeit vor allem von Ohno (1970) entwickelt worden sind. Goldschmidt kommentierte die Frage u.a. wie folgt (p.482):
Gene verändern sich nur durch Mutation in eine oder mehrere Allelformen, die im Grunde genommen alle die gleiche Wirkung haben. Alle Allele des scute-Locus verändern das Borstenmuster und alle Allele des white-Locus die Augenfarbe. Noch nie hat jemand ein scute-Allel gefunden, das einen Einfluss auf die Augenfarbe ausübt. Wenn wir die Pseudoallele als verdoppelte Gene ansehen, dann würde diese Theorie voraussetzen, dass sie verschieden werden. In allen uns bekannten Fällen hat das angenommene Duplikat im wesentlichen die gleiche Wirkung und ist nur in Kleinigkeiten verschieden, verschieden jedoch nur in dem Ausmaß, in dem sich ein Allel von einem anderen Allel unterscheidet. In der Tat sind Pseudoallele stets zunächst als gewöhnliche multiple Allele beschrieben worden, die darüber hinaus ihre eigenen Allele mit ähnlicher Wirkung hervorbringen. Was hier an Verschiedenheiten vorkommt, liegt im Rahmen der Wirkung multipler Allele.
- Worauf auf eine Gruppe von white-Allelen bei Drosophilahingewiesen wird. Auf der Seite 483 fährt der Autor fort:
Diejenigen, die glauben, dass durch Verdoppelung neue Gene entstehen, sagen, bei der Pseudoallelie sähen wir nur den Beginn der Entstehung neuer Gene. Nach einer Million Jahre werde eines von ihnen ein Gen für die Bildung einer neuen, heute noch völlig unbekannten Struktur sein.
Und er schneidet kurz darauf ein weiteres entscheidendes Problem an, wenn er fortfährt:
Wie kann das neue Gen aus einer Verbindung mit aufeinanderfolgenden Schritten eines Synthesevorganges herausgelöst werden, um eine vollständig neue Reaktionskette zu katalysieren, wie wir annehmen müssen, wenn ein völlig neues Gen für ganz neue Evolutionsschritte entsteht?
Ohno hat in seiner Monographie EVOLUTION BY GENE DUPLICATION (1970) diese Probleme nicht lösen können und ist im Grunde genommen nicht wesentlich über Bridges Ansatz von 1918 hinausgekommen, wenn er im Kapitel XIII "The Creation of a New Gene from a Redundant Duplicate of an Old Gene" zum Beispiel schreibt (p.72):
Natural Selection would ignore the redundant locus, and thus it is free to accumulate a series of forbidden mutations, aided by intragenic recombination. As a result, the polypeptide chain specified by it might finally aqcuire a function which is quite different from that assigned to the original gene. In such a way a series of new genes with previously non-existent functions must have emerged during evolution. The creation of a new gene from a redundant copy of an old gene is the most important role that gene duplication played in evolution.
(Zum Thema "forbidden Mutations" vgl. unsere Arbeit pp. 349 und 360). Der einzige Fortschritt gegenüber früheren Arbeiten lag darin, dass Ohno jetzt eine vergleichende Betrachtung verschiedener Aminosäurensequenzen verwandter Stoffklassen (Chymotrypsin-Trypsin, Hämoglobin
a, b, g, e -Ketten etc.) mit klaren Homologien aufführen konnte, die die Hypothese der Genduplikation für die Entstehung neuer Gene konkretisieren und damit interessanter machten.Schmidt kommentiert 1985, p.63, das Beispiel der Hämoglobine wie folgt:
Als Paradebeispiel für die Neuentstehung von Genen durch Genduplikation eignen sich die Gene für verschiedene Hämoglobine schon allein deshalb nicht, weil hier - wie in den meisten anderen Fällen auch - die homologen Proteine die gleiche Funktion aufweisen. Nach OHNO soll jedoch die Genduplikation vor allem die Entstehung von Genen mit neuen Aufgaben erklären. Das ist ein grundlegender Unterschied.
Gerade die von Ohno und allgemein in Lehrbüchern aufgeführten Beispiele verdeutlichen Goldschmidts Kommentare und Einwände zu diesem Thema, insbesondere die Frage, wie 'das neue Gen aus einer Verbindung mit aufeinanderfolgenden Schritten herausgelöst werden kann, um eine vollständig neue Reaktionskette zu katalysieren' bzw. Glied einer neuen Reaktionskette zu werden.
Um auf Ohnos oben zitierte Gedanken zurückzukommen: Die Polypeptidkette sollte nach zahlreichen Mutationen und intragenischer Rekombination schließlich eine im Vergleich zum ursprünglichen Gen ganz andere Funktion erwerben, und auf diese Weise soll eine Serie neuer Gene mit vorher nicht-existenten Funktionen aufgetreten sein. Wie oben schon zitiert, bringt ein einzelnes neues Gen dem Organismus in der Regel noch gar nichts, weil für die Synthese neuer Verbindungen meist Genwirkketten notwendig sind (vgl. p 233), wobei die Frage nach der Regulation und Korrelation mit dem Plasma noch gar nicht miteinbezogen ist. Selbst wenn wir die unwahrscheinliche Entstehung einzelner neuer Gene mit (potentiell) ganz anderen Funktionen als gegeben voraussetzen, würde - mangels sinnvoller Expression und stabilisierender Selektion - das neue Funktionspotential durch weitere Mutationen wieder verlorengehen und somit nichts erreicht werden. Und dazu sei noch einmal betont, dass die potentiell neuen Genfunktionen sich auch sinnvoll in dem gegebenen Gesamtsystem eines Organismus einfügen müssten. Was nützte jedoch z.B. die Bildung von Rhodopsin im Kambium einer Kastanie (- um das Problem einmal plastisch darzustellen) oder die Synthese von Chlorophyll in der Magenschleimhaut eines Elefanten oder der Aufbau von Chitin bei der Spindelbildung der Mitose?
Ohno hat die Problematik 1985, p.160, wieder aufgegriffen und er schreibt, dass nach Genduplikation für die redundanten Kopien der Gene zwei Möglichkeiten für weitere Veränderungen bestehen:
While being ignored by natural selection, they are free to accumulate random base substitutions, deletions and insertions. Consequently a few may emerge triumphant as new genes endowed with somewhat novel functions. However, a far more likely fate is degeneracy due to the loss of promoter sequence, premature chain terminations, frameshifts etc. For every new gene that emerged triumphant, tens or hundreds of redundant copies must have joined the ranks of junk DNA. Indeed the abundance of pseudogenes (recent degenerates) attests to the inefficacy of gene duplication as a means of acquiring new genes with novel functions (Kursiv vom Verfasser.) (Vgl. auch Nachtrag p.545. Nach Behauptung: "A gene with a new function generally evolves from a duplicate gene, ..." setzt auch Nei 1987, p.409 hinzu: "... but duplicate genes often become nonfunctional by deleterious mutations.")
In der Arbeit von 1985 begründet Ohno ausführlich, dass wegen der im Vergleich zu Bakterien genaueren Arbeitsweise der DNA-Polymerasen und der verfeinerten Reparatursysteme auch funktionslose Gene noch lange Zeit im Genom von Säugetieren erhalten bleiben (p. 163):
The inevitable conclusion to be drawn is that, contrary to the generally held belief, those genes that become useless, and therefore dispensable, would not readily disappear from the genome of mammals (in particular) and vertebrates (in general). Instead they will linger for millions of years.
Abgesehen von der im 1. Zitat gebrauchten pathetischen Ausdrucksweise von "triumphierend entstehenden" neuen Genen, scheint der Autor inzwischen doch etwas vorsichtiger mit seinen Hypothesen zur Gen-Duplikation geworden zu sein. War es 1970 noch "eine Serie neuer Gene mit vorher nicht-existenten Funktionen, die (auf diese Weise) in der Evolution entstanden sein müssen", so sind es jetzt nur noch ein paar neue Gene "with somewhat novel functions".
Das ergibt sich zum Teil aus der Schlussfolgerung, dass funktionslose Gene noch Millionen von Jahren im Genom der Wirbeltiere (und wohl Eukaryonten überhaupt) erhalten bleiben können.
Li schreibt 1980, p.256:
There is no general agreement as to how a new gene may emerge from a redundant copy. OHNO (1973) believes that in order to avoid degenerating into a worthless DNA sequence, a redundant copy must acquire a new function beneficial to the organism and come under the protection of natural selection. However, KOCH (1972) and RIGBY, BURLEIGH and HARTLEY (1974) think that the redundant copy should go through a "silent" period in order to allow time and opportunity for accumulating a sufficient number of mutations. In my view, the second possibility is less likely. Degeneracy usually results from a frameshift mutation or a chain-terminating nonsense mutation. In the former case the chance for the degenerate gene to become functional again is obviously extremely small. In the latter case, back mutation is possible but, before it occurs, additional deleterious mutations may have accumulated since a silenced gene is no longer protected by natural selection.
Zur Unterstützung der These Ohnos weist Li darauf hin, dass bei Bakterien Änderungen in der Substratspezifität durch wenige Mutationen erreicht werden können. Abgesehen davon, dass wegen Beweismangels sowieso keine Einigung unter den Autoren erzielt worden ist, würde das letztere generell eine relativ rasche Evolution mit wenigen Aminosäurerest-Substitutionen bedeuten (Hypothese vom schnellen Funktions-Neugewinn zur Vermeidung der Degeneration). Wir kommen jedoch weder mit den Millionen Jahre im Genom verbleibenden funktionslosen Genen noch mit einer relativ schnellen Evolution der Gene zu neuen Funktionen weiter: in beiden Fällen wird eine tiefgreifende Umkonstruktion der Gene unwahrscheinlich (ich erinnere an Goldschmidts Einwand, dass die bekannten Gen-Duplikate voneinander nicht verschiedener sind als Allele des selben Gens), und der Aufbau neuer Genwirkketten bleibt mit diesem Ansatz in jedem Falle unverständlich.
Weiter ist hervorzuheben, dass trotz verfeinerter Reparaturmechanismen etc. bei den Eukaryonten die funktionslosen Gene wesentlich schneller degenerieren werden als die bedingt redundanten Sequenzen des übrigen Genoms (selbst wenn noch nach größeren Zeiträumen Homologien nachweisbar sind). Die Degeneration führt jedoch nicht zu Funktionsgewinn, sondern zu Nonsense-Sequenzen. Für den Aufbau neuer Gene gelten die Seite 359 vorgelegten Wahrscheinlichkeitsberechnungen.
Wäre es anders, dann müssten die polyploiden Arten, bzw. diejenigen mit dem größten Anteil an repetitiven Genen alle anderen längst überflügelt haben. Das ist nicht der Fall.
Hoyle und Wickramasinghe bemerken zur Frage der Tandem-Duplikationen 1981, pp. 100/101:
It is possible that tandem duplication of one or several genes could produce a marked increase in the amount of genetic material over only a few thousand generations, but it is doubtful that any marked functional diversity could arise in this way. Indeed, quite the reverse. In writing about the lungfish, S. Ohno remarks:
By establishing such a system [tandem duplication] the organism effectively forfeited an opportunity for further evolution. In a manner of speaking, the genome became frozen, while containing enormous genetic redundancy. It is clear that in doing so such a lineage reached an evolutional dead end. It will be shown that what happened to the lungfish also happened to salamanders and newts… Indeed, this side branch stopped dead at the amphibian stage.
… At all events, tandem duplication does not solve the evolutionary dilemma. It might give a rapid increase in the quantity of genetic material, but it only does so by being highly repetitive, and this will not give a sequence of 'quantum jumps' in the forms of plants and animals, such as is needed to provide for the divergent evolutionary branches...Repetitions will give some changes, of course, by altering the quantities of certain proteins, but, as Ohno remarks in the above quotation, the changes are much more likely to be stultifying than to lead to adventurous new possibilities.
(Zum Pflanzenreich vgl. Stebbins, zitiert p.212)
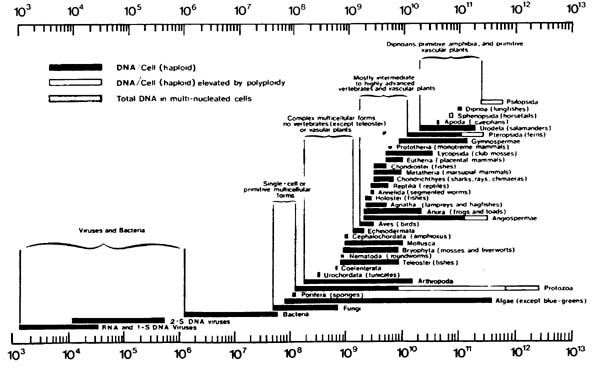
Wäre die Anzahl der Duplikationen mit der Differenzierungshöhe korreliert, so müssten die höher organisierten Lebensformen höhere Duplikationszahlen aufweisen als die niederen. Das trifft ebenfalls nicht zu. Die höchsten Werte erreichen die Sphenopsida, Dipnoa und Psilopsida (vgl. die Abb. 56).
Darüber hinaus wurde pp. 406/407 dokumentiert, dass Spezialisierung auch innerhalb mancher Gruppen mit einer Verringerung des DNA-Gehalts verbunden ist (allerdings lassen sich Höherdifferenzierung und Spezialisierung nicht ohne weiteres gleichsetzen, so dass dieser Punkt nicht auf alle zitierten Gruppen zutrifft).
In vielen Lehrbüchern ist jedoch schon alles vollkommen klar:
"Duplikation ist die Verdoppelung eines DNA-Abschnittes in gleicher Orientierung (Tandem) oder inverser Lage (Palindrom) auf Kosten des homologen Chromosoms. Vermutlich sind so die weitverbreiteten "repetitiven Sequenzen" der DNA entstanden, aber auch neue Gene im Laufe der Evolution, indem bei Erhaltung der alten Information die verdoppelten Bereiche störungsfrei über Punktmutationen neue Polypeptide zur Erprobung bereitstellten:
Die Hämoglobin-Ketten sind durch mehrfache Duplikationen eines ursprünglich ca. 210 Nukleotidpaare umfassenden Vorläufer-Gens entstanden" (Vogel und Angermann 1984, p.477).
"Duplikationen sind die Grundlage für die Entstehung neuen genetischen Materials und haben daher eine große Bedeutung für die Evolution" (Günther 1984, p.243).
Beispiele gibt es dafür auch aus der Primärliteratur (Gosalvez et al. 1980, Sharma 1981, Session 1982, Hill et al. 1985 u.v.a.).

Abb. 56: The range of nucleotide content in major taxonomic categories. (Aus Hoyle und Wickramasinghe 1981 nach B. John und K. R. Lewis.)
Schmidt hat 1985, pp. 60-73 eine gründliche Kritik zum Thema der Genduplikationen vorgelegt, die unsere bisherigen Ausführungen weiter vertiefen. Ich möchte einige längere Passagen aus diesem Abschnitt zitieren:
Die Tatsache, daß es Proteingroßfamilien gibt - zu denen man teilweise auch Proteine zählt, die in weniger als 50 % ihrer Aminosäuren übereinstimmen - beweist selbstverständlich ebenfalls in keiner Weise, daß die zugehörigen Gene aus einem Gen entstanden sind. Auch hier gilt, was bereits gesagt wurde: Wenn eine bestimmte Erbinformation sich nur durch eine Nucleotidsequenz ausdrücken läßt, dann muß diese zwangsläufig gewählt werden. Nicht jede zufällige Nucleotidsequenz ergibt einen "Sinn". Wahrscheinlich sind auch hier Gesetzmäßigkeiten am Werk, die wir noch nicht durchschauen. Niemand dürfte aus der Tatsache, daß sich z.B. die beiden Worte "gehen" und "hegen" aus den gleichen Buchstaben zusammensetzen, den Schluß ziehen, daß diese beiden Begriffe oder Tätigkeiten auf eine gemeinsame Wurzel zurückgehen.
Auch für das Hämoglobin ist die Situation keineswegs so leicht durchschaubar, wie es auf den ersten Blick scheint. Die Gene der einzelnen Hämoglobinketten wurden nämlich bei der Duplikation durch Translokation verlagert und liegen nicht nebeneinander. Man sollte deshalb eigentlich annehmen, daß nunmehr zwei selbständige Hämoglobinmoleküle gebildet werden. Das ist jedoch überraschenderweise nicht der Fall. Die beiden getrennt synthetisierten bzw. codierten Hämoglobinketten vereinigen sich nachträglich zu einem einzigen Protein. Das gleiche gilt für die verschiedenen Ketten der Immunglobuline. Auch das spricht nach meiner Ansicht gewichtig dafür, daß selbst bei diesen Paradebeispielen der Genduplikation nicht nur der Zufall in Form von Mutationen beteiligt war. Offensichtlich wirken auch hier noch unbekannte Gesetzmäßigkeiten mit, die wir global als Genregulation bezeichnen. Diese stellt - wie immer man sie letztlich interpretieren mag - eine gerichtete Steuerung dar, die uns noch ausführlich beschäftigen wird.
Wenn demnach ein bestimmtes Protein zwei Polypeptidketten aufweist, die auf eine Genduplikation hindeuten, so sollten wir uns hüten, diese Verdoppelung vorschnell als Zufall oder gar als zufällige Mutation abzutun, weil es durchaus sein könnte, daß diese Duplikation aus funktionellen Gründen nicht minder notwendig ist als eine beliebige andere strukturelle Besonderheit.
Da die Neuentstehung von Genen durch Genduplikation mit nachfolgenden Mutationen des Reserve-Gens nichts anderes ist als eine Variante der mutativen Abänderung von Genen generell, liegen auch die Haupteinwände dagegen auf der gleichen Ebene:
Nehmen wir an, ein dupliziertes Gen, das als Ausgangsmaterial für ein Gen mit neuer Funktion dienen soll, enthielte 1.000 Einzelnukleotide. Nehmen wir weiter an, das neue Gen würde sich im Endzustand von dem duplizierten Ausgangsgen in nur 10 Einzelnukleotiden - also nur in 1 % unterscheiden und Letalmutationen wären nur zehnmal häufiger als Gewinnmutationen: Selbst in diesem besonders günstig gelagerten Fall, der im Durchschnitt sicher nicht als Regel betrachtet werden kann, hätte das neue Gen aus folgenden Gründen nur eine praktisch vernachlässigende Chance, auf dem Wege von Mutationen gebildet zu werden:
1. Die Wahrscheinlichkeit, daß eine zufällige Mutation eines der 10 zu verändernden Nukleotide betrifft, ist durchschnittlich 99mal niedriger, als daß eines der 990 Nukleotide mutiert, die unverändert bleiben sollen. Dabei ist noch nicht einmal berücksichtigt, daß es ganz verschiedenartige Mutationen gibt und daß für das neue Gen nur ein ganz bestimmter Typ - nämlich eine Punktmutation - in Frage kommt.
2. Diese erste Punktmutation müßte an spezifischer Stelle zum Austausch einer Aminosäure durch eine andere führen, und zwar durch eine ganz bestimmte Aminosäure. Da es 20 Aminosäuren gibt, ist es 19 mal wahrscheinlicher, daß nicht die richtige, sondern eine falsche Aminosäure eingesetzt wird. Nehmen wir eine Mutationsrate von 10-5 an, die sehr wohlwollend zugunsten des Neodarwinismus bemessen ist, da auf Punktmutationen - wie bereits erwähnt - sicher nur ein Teil dieser Mutationsrate entfällt: Dann erhöht sich die Unwahrscheinlichkeit einer "richtigen" Mutation bereits dadurch auf 99 x 19, also auf fast das 2.000fache. Die Wahrscheinlichkeit dieser Mutation wäre dann 10-5 x 99 x 19 = nahezu 1 : 200 Millionen. Schon für die erste erforderliche Aminosäuresubstitution ist also eine nachteilige Mutation ungleich wahrscheinlicher.
3. Es ist nicht anzunehmen, daß bei 10 notwendigen Aminosäurenaustauschen für das neue Gen schon die erste Punktmutation - selbst wenn dadurch die richtige Aminosäure codiert wurde - zu einem Selektionsvorteil geführt hätte,
a) weil damit erst 10 % der für das neue Gen zu fordernden Veränderungen realisiert gewesen wären,
b) weil Mutationen in aller Regel ohnehin - zum Glück für die Betroffenen rezessiv auftreten. Da die Selektion am Phänotyp angreift, ist zudem schon aufgrund der Rezessivität kein Selektionsvorteil zu erwarten. Das neue Gen hätte demnach günstigstenfalls Aussicht, für einige Zeit mitgeschleppt zu werden, nicht aber, sich mit Hilfe der Selektion durchzusetzen. Wie wir auf S. 41 gesehen haben, wäre in diesem Falle schon in der 2. Generation in fast 50 %, und nach 13 Generationen in 90 % der Fälle mit dem Verschwinden des mutierten Allels zu rechnen.
4. Die zweite notwendige Punktmutation müßte durch einen weiteren Zufall - diesmal schon von fast unmeßbarer Unwahrscheinlichkeit bei einem direkten Nachkommen des Trägers der Erstmutation auftreten. Die Unwahrscheinlichkeit dieses Ereignisses ist deshalb so besonders groß, weil - wenn überhaupt - zu diesem Zeitpunkt nur noch einzelne Merkmalsträger der Erstmutation als Nachkommen vorhanden wären. Selbst wenn wir den günstigsten Fall annehmen, daß die erste Punktmutation mit keinerlei Nachteilen für den Träger verbunden war und damit die Zahl seiner Nachkommen nicht reduziert wurde, reicht schon die fehlende Möglichkeit einer Vermehrung der Nachkommenzahl mit Hilfe der Selektion aus, um die Zahl der potentiellen Anwärter für die zweite Punktmutation auf ein Minimum zu reduzieren.
Als Punkt 5 weist Schmidt darauf hin, dass bei geographischer Isolation Homozygotie im zuerst mutierten Allel auftreten und das bislang unveränderte Allel gänzlich eliminiert werden könnte. Im Vergleich zum bewährten Allel müsste man jedoch mit schwerwiegenden Selektionsnachteilen rechnen, was nun auch keine solide Basis für weitere Mutationen wäre.
6. Man kann darüber diskutieren, ab der wievielten Mutation theoretisch ein Selektionsvorteil für das neue Gen, das durch Duplikation entstehen soll, zu erwarten ist. Sicher dürfte dies vor allem davon abhängen, ob die Mutationen in einem prospektiven Wirkungszentrum erfolgte oder nicht. Da die eigentlichen Wirkungsgruppen meist nur einen kleinen Teil des gesamten Gens ausmachen, muß man wohl davon ausgehen, daß ein Selektionsvorteil in der Regel frühestens dann erwartet werden kann, nachdem zumindest ein wesentlicher Teil der 10 nötigen Punktmutationen an dem Tandem-Gen erfolgte. Da aber auch in diesem Falle nicht die Substitution einer beliebigen Aminosäure im Wirkungszentrum genügt, ist auch hier die Wahrscheinlichkeit verschwindend gering, daß ausgerechnet diejenige der 20 Aminosäuren, die benötigt wird, an die richtige Stelle kommt, bevor "falsche" Substitutionen an anderer Stelle stattgefunden haben.
7. Es liegt auf der Hand, daß die Wahrscheinlichkeit zur Neuentstehung eines Gens auch auf dieser Basis umso geringer ist, je weniger überlebende Nachkommen der oder die Träger der Genduplikation und der mutierten Tandem-Gene hat bzw. haben: ob es sich um ein mutativ zu veränderndes Tandem-Gen oder um ein nicht dupliziertes Gen handelt, das durch Mutationen in analoger Weise abgeändert werden soll, spielt dabei keine Rolle. Es wäre demnach zu erwarten, daß das Angebot an Mutationen - und damit die Evolutionsgeschwindigkeit der betreffenden Art - umso größer ist, je mehr Nachkommen eine Art hervorbringt und je kürzer ihre Generationszeit ist. In Wirklichkeit ist jedoch das genaue Gegenteil der Fall, wie auf S. 45 gezeigt wurde: Die Evolutionsgeschwindigkeit nimmt mit der bereits erreichten Evolutionshöhe zu.
8. Wie immer wir die Dinge betrachten: Mit jeder weiteren, für die Neuentstehung eines Gens notwendigen Punktmutation wächst ihre Unwahrscheinlichkeit nach den Gesetzen der geometrischen Reihe. Da - wie in Punkt 3 gezeigt wurde - zumindest für die ersten Mutationsschritte im Regelfalle nicht auf die Hilfe der Selektion gehofft werden kann, bietet nach meiner Ansicht auch die Genduplikation keine Möglichkeit, denkbar unwahrscheinliche Zufälle wenigstens einigermaßen wahrscheinlich zu machen...
Der neunte Punkt, dass Genduplikationen nur eine Sonderform unter vielen anderen Mutationstypen und nachgewiesene mutative Genduplikationen insgesamt so selten seien, dass sich hierfür nicht einmal die Mutationsraten angeben ließen, ist inzwischen zu korrigieren (siehe unten).
Unter Punkt 10 schreibt der Autor unter anderem:
Bei der Genduplikation kommt - im Gegensatz zu Punktmutationen - noch hinzu, daß duplizierte Gene zweifellos die Chromosomenpaarung homologer Abschnitte bei der Mitose und Meiose erschweren und so zusätzlich zu Störungen in deren Ablauf führen können, die ebenfalls weit eher Selektionsnachteile erwarten lassen als einen selektiven Nutzen.
Homologe Proteine können nicht als Argument für ein ausreichend langes Verweilen im Genpool gelten, weil sie im Regelfalle eine bereits vorhandene Genfunktion nur modifizieren, nicht aber zu grundlegend neuen Eigenschaften führen. Die Hauptfunktion des Hämoglobins etwa - die Übertragung von Sauerstoff - blieb von der Entstehung neuer Ketten gänzlich unberührt; Hämoglobin blieb Hämoglobin, auch wenn einige Aminosäuren durch andere ersetzt wurden.
Weitere Punkte beim Verfasser.
Die oben dokumentierte und häufig anzutreffende Lehrbuchaussage zur Bedeutung der Gen-Duplikationen ist eine evolutionistische Deduktion, die sich der empirischen Reproduktion bedauerlicherweise entzieht. Und ausgerechnet die immer wieder als Musterbeispiel zitierte X-chromosomale Bar-Duplikation von Drosophilawirkt sich negativ aus, indem mit dieser und jeder weiteren Duplikation der Bar-Region das Facettenauge schrittweise auf wenige Facetten reduziert wird:
"...the
dominant mutation Bar in Drosophila produces a slitlike
eye
instead
of
the normal oval one. Cytological study of the polytene chromosomes have
shown the Bar mutation on the X chromosome is, in fact, a tandem
duplication that probably results from an unequal
crossover...: Gametes containing the deletion presumably die or
produce inviable zygotes. Gametes containing the duplication, however,
produce hemizygous male offspring with severely reduced eyes or
heterozygous female offspring with slightly reduced eyes.
Evidence
that asymmetric pairing and crossing-over produces higher oders of
duplication comes from studying homozygous Bar
females. Occasionally,
such females produce offspring with extremely small eyes called
"double bar". Each offspring is found to carry three doses of the
Bar
region in tandem" (Hinweis auf Abb.) (Griffith et al.: An Introduction to
Genetic Analysis 1993, p. 216; bold von
mir).
Die Gruppe von Beispielen, zu der die ebenfalls viel zitierte Methotrexate-Resistenz durch Amplifikation des für die Dihydrofolate Reduktase (DHFR) kodierenden Gens gehört, wird von S.L.Wolfe wie folgt kommentiert: "In tumor cells, for example, development of resistance to anticancer drugs has in some cases been traced to chance amplification of genes coding for the production of enzymes that break down the drug, or to transport proteins that promote excretion of the drug from the cell. Although beneficial to the survival of the cancer cells, the random amplification producing drug resistance is less lucky for the individual harboring the tumor" (Wolfe 1993, p. 980; kursiv von mir). Mit anderen Worten können letztlich beide an der Amplifikation zugrundegehen!
Nach einer umfangreichen Studie von J. Gressel (1985) wird die (zum Teil auch auf Gen-Duplikationen beruhende) Herbizidresistenz von Kulturpflanzen und 'Unkräutern' in aller Regel mit einer herabgesetzten Fitness der resistenten Populationen bezahlt ("Haldane was right; there is a consistent loss of fitness upon selection for herbicide resistance" - Details siehe ARTBEGRIFF, pp. 380-383). Wird nach einiger Zeit der Herbizideinsatz beendet, so kehren die Populationen regelmäßig unter Abbau der Resistenz zu ihrem ursprünglich höheren Funktionsniveau zurück (Gesetz der rekurrenten Variation).
Sieht man sich konkrete Beispiele für Duplikationen in der Primärliteratur an, so findet man z.B. folgende interessante Tatsachen:
Welker et al. berichten 1982, p.717, dass bei der von ihnen an Dictyostelium discoideum untersuchten Duplikation in allen Fällen die Duplikationslinien langsamer wuchsen als die meisten normalen, d.h. die Duplikation nicht aufweisenden Linien. "The presence of the duplication had adverse gene dosage effects on colonial growth and development" (p.719). Die Duplikation wurde in hoher Frequenz durch 254 nm UV-Bestrahlung von Zellsuspensionen "kuriert" (Formulierung von den Verfassern).
Batterham et al. gehen 1984 auf den Ursprung und die Expression der Duplikation eines Alkoholdehydrogenase-Gens bei verschiedenen Drosophila-Wildpopulationen ein und stellen u.a. fest:
(p. 652:) The existence of the duplication is ...not explained by any simple hypothesis invoking an advantage due to increased enzyme activity. Similar levels of activity and similar larval-adult ratios are found in species with and without the duplication and from diverse habitats.
(p. 655:) One consequence of a duplication can be to increase the amount of gene product which could elevate enzyme activity. This does not appear to be the case with the duplication for Adh in the mulleri subgroup since specific activities are generally lower than comparable species without the duplication. It is possible that a duplication can provide a means of reducing the level of enzyme activity during certain life stages (i.e., adult) yet maintaining sufficient levels at other stages (i.e., larvae). However ... species without the duplication can also have reduced adult levels.
Die Autoren setzen die Entstehung dieser Duplikation für das Miozän (also nach gängiger geologischer Zeitrechnung vor 5 bis 26 Millionen Jahren) an. Das würde für Ohnos These von der langen Verweildauer nutzloser Gene sprechen.
Roberts und Broderick haben bei Drosophila melanogaster 33 röntgenstrahleninduzierte Tandem-Duplikationen beschrieben. Eine signifikante Reduktion der Fertilität konnten sie bei den für kurze und mittellange Duplikationen Heterozygoten nicht feststellen (Absinken der Fertilität erst bei 69 Extrabanden der 3R Duplikationen und bei 206 der 2L-Region). Zu den homozygoten Duplikationslinien bemerken die Verfasser 1982, p.83:
Most of the duplications recovered here could not be studied as homozygotes owing to recessive lethality (recessive lethals are often introduced in and near rearrangements induced by high X-ray fluxes and are separated from crossover suppressors with difficulty), or inability to insert nearly markers owing to strong crossover suppression.
Solche Letalfaktoren dürften in Verbindung mit Duplikationen in verringertem Maße auch bei 'spontanen' Mutationsereignissen in der Natur auftreten (vgl. dazu einige der auf den Seiten 343-345 aufgeführten natürlichen mutagenen Agenzien).
Die Wirkung duplizierter Abschnitte eines Chromosoms auf die Crossing-over-Häufigkeit kann sehr unterschiedlich sein. In den vorliegenden Beispielen war sie im heterozygoten Zustand stark herabgesetzt und konnte in den meisten Fällen wegen der Letalfaktoren nicht in homozygoten Linien untersucht werden.
Die Verfasser weisen jedoch darauf hin, dass nicht alle Tandem-Duplikationen das Crossing-over reduzieren. So erhöhen kurze X-Duplikationen (8-18 Banden) im homozygoten Zustand die Crossing-over-Häufigkeit; in diesen Fällen war jedoch die Rekombination schon im heterozygoten Zustand unverändert.
Nach Spofford 1972 ist die Dissoziationsrate duplizierter Gene allein schon durch Crossing-over wesentlich höher als die Mutationsrate, die zu neuen Genen mit Selektionsvorteil führen könnte. Er sieht sich deshalb veranlasst, zur Rettung der Genentstehung durch Duplikationen ein Modell zu entwerfen, in dem die monogene Heterosis eine besondere Rolle spielen soll: Durch Verdoppelung eines heterotischen Gens seien größere Verbreitungschancen in einer Population und damit eine größere Wahrscheinlichkeit der Entwicklung neuer Funktionen gegeben. Ähnlich wird die Entstehung neuer genetischer Informationen von Alberts et al. 1983, pp. 775/776 erklärt:
... the new genes can become common through heterozygote advantage without loss of the original genes; and, finally, they can be inserted as additions to the genomes through gene duplication and genetic recombination.
Zum Thema Heterosis vgl. die Ausführungen pp. 383-392. Genau genommen multiplizieren sich hier die Unwahrscheinlichkeiten. Wie ich im Detail gezeigt habe, handelt es sich bei der monogenen Heterosis um extrem seltene Erscheinungen unter extremen Umweltverhältnissen. Dieses Phänomen als Regel für die Entstehung neuen genetischen Materials und für den Ursprung der Lebensformen aufzubauen, widerspricht allen bekannten Tatsachen.
Warum, fragt man sich zum Beispiel, ist Genverdoppelung mit anschließender Rekombination bei der durch Heterozygotie in den Genen für Hämoglobin S und normales Hämoglobin bedingten Malariaresistenz nicht längst die Methode gewesen, um den fürchterlichen Verlust bis zu 25 % der Nachkommenschaft in malariaverseuchten Gebieten zu vermeiden, der allein durch die homozygoten S-Träger bedingt ist? Und dazu kommen noch die 25 % nicht malariaresistenten Nachkommen, von denen viele durch den Malaria-Parasiten Plasmodium falciparum vorzeitig sterben. Der ungeheure Selektionsvorteil von über 25 % höherer lebensfähiger Nachkommenschaft müsste die weite Verbreitung solcher Gen-Duplikationen unter diesen extremen Verhältnissen in relativ kurzen Zeiträumen garantieren. Wenn sich das Sichelzellgen über große Teile Afrikas verbreiten und in hoher Frequenz auftreten konnte (bis zu 44 % HbS-Heterozygote bei Westafrikanern), dann sollte bei Richtigkeit des Duplikationsansatzes in Verbindung mit Heterosis auch weitgehende Resistenz durch Genduplikation und Rekombination in den nach evolutionistischen Voraussetzungen angesetzten großen Zeiträumen schon vielfach aufgetreten sein und sich anstelle des bestehenden verlustreichen Polymorphismus längst durchgesetzt haben.
Nach Roberts und Broderick 1982, p. 85, ist die Frequenz des Auftretens neuer Tandem-Duplikationen höher als bisher angenommen. Sie ist nach Vergleich mit den Ergebnissen von Gelbart und Chovnick 1979 (Duplikationsrate von 1 x 10-5 am rosy Locus von Drosophila) mit den eigenen Befunden "some 20 times the average mutation rate of a specific locus". Fast alle von den Autoren festgestellten Duplikationen waren mit einer Deletion in oder nahe den betreffenden Loci verbunden waren. Die Gen-Duplikationsrate beträgt nach Roberts und Broderick bei Drosophila 2 x 10-5.
Aber gehen wir einen Schritt weiter: Nehmen wir einmal an, die Duplikation hätte sich wiederholt ereignet und durchgesetzt. Die Afrikaner wären weitgehend malariaresistent. Sie wären jedoch auch alle schwach anämisch und hätten im Gegensatz zu Populationen aus nichtmalariaverseuchten Gebieten mit folgenden weiteren Schwierigkeiten zu kämpfen, die McLeod 1982, p.21 für heterozygote Träger des Sichelzellmerkmals wie folgt beschrieben hat (SCT = Sickle Cell Trait):
The following is a list of clinical manifestations attributed to SCT; however, many individuals who are trait carriers may live totally unaware of their condition. Carriers have been shown to have greater risks with local or general hypoxemia in general anesthesia, pulmonary infarction at both high altitude and at sea level, splenic infarction, massive hematuria (bloody urine), renal papillary necrosis, renal medullary necrosis, renal failure, retroperitoneal fibrosis, higher incidences of leg ulceration, lower birth weight in infants of SCT mothers, retinal arterial occlusion following ocular trauma, and significant increased associations between maternal SCT carriers and perinatal mortality when mothers were subjected to anoxic stress.
InSickle Cell Hemoglobinother impairments are mentioned. "Aseptic femoral head necrosis, superior longitudinal sinus thrombosis, and serious ocular pathology." Carriers are reported to have higher incidences of Salmonella infections. Although SCT may infer some protection against leprosy, it also predisposes carriers to acute exudative tuberculosis. There appears to be a high correlation between pulmonary emboli, oral contraceptives, and SCT. And following minor leg infections, carriers often have recurrent ulcers because of impaired healing. Dr. David A. Sears writing in the American Journal of Medicine includes increased abnormalities within SCT carriers for splenit infarction, bacteriuria, phelonephritis in pregnancy, and in his discussion adds, "Even those who minimize the risks of AS hemoglobinopathy suggest that certain real hazards do exist for carriers of the trait.''
In malariafreien Gebieten nimmt folglich der Prozentsatz der Heterozygoten wieder ab. Bei den von Westafrikanern abstammenden schwarzen US-Bürgern ist er in malariafreien Zonen auf 9 % abgesunken (nach Vogel und Angermann 1984, p. 501). Unter den extremen Verhältnissen einer stark malariabetroffenen Umwelt sollte jedoch der SCT-Selektionsvorteil die Nachteile weiter deutlich überwiegen und die oben zitierte Duplikationshypothese zutreffen. Das ist jedoch nicht der Fall.
Harris hat 1980 in seiner zusammenfassenden Arbeit einen guten Überblick über beim Menschen in den hämoglobinkodierenden DNA-Sequenzen aufgetretenen unequal Crossing-overs und entsprechenden Sequenzveränderungen gegeben. Es ist bisher nicht möglich, im Vergleich zu den vorliegenden normalen Sequenzen auch nur ein einziges im Sinne der Selektionstheorie positives Beispiel für Gen-Duplikationen beim Menschen vorzulegen. Im Gegenteil, die in Form von Genfusionen vorliegenden duplizierten Sequenzen scheinen sich spätestens im homozygoten Zustand regelmäßig negativ auf die betroffenen Menschen auszuwirken (z.B. Anti-Lepore-Hämoglobine). Dass im fein ausbalancierten physiologischen Getriebe Zusatzketten eine Störfunktion haben könnten, versteht sich von selbst. Erst wenn sie in Form nicht mehr exprimierter Pseudogene vorliegen und dabei nicht die Regulation der bisherigen genetischen Information beeinträchtigen würden, könnte man von solchen Genen allgemein als neutralen Sequenzen sprechen. Die Hoffnung jedoch, dass dann durch Punktmutationen eine verbesserte neue Funktion aufgebaut und das neue Gen auch zur rechten Zeit wieder "angeschaltet" würde, ist nach den oben aufgeführten Wahrscheinlichkeiten und empirischen Befunden nicht überzeugend. Aber selbst wenn wir einmal annehmen, dass sich die Heterosishypothese in Verbindung mit Gen-Duplikationen im konkreten Fall der Malariaresistenz vieler afrikanischer Populationen als richtig erwiesen hätte, würden nicht einmal extreme 'Splitters' unter den Systematikern solche Populationen als eigene Menschenarten einstufen.
Obwohl man dem Gedanken der Evolution durch Gen-Duplikation in der neueren Literatur immer wieder begegnet, geht keines der konkreten Beispiele über die von Goldschmidt beschriebenen Grenzen hinaus. (Vgl. jedoch Nachträge pp. 558-566).
Die Genfamilien selbst verlangen jedoch eine Erklärung.
Die Spezifität der Sequenzen (bei der schon die Substitution einer einzigen Aminosäure homozygot tödlich sein kann), das Zusammenspiel auf genregulatorischer, physiologischer und organismischer Ebene in Korrelation mit der Umwelt und in genauester Abstimmung mit Tausenden anderen Sequenzen und Funktionen bieten Beispiele für Synorganisation, die auch synorganisiert ins Dasein gekommen sein müssen. D.h. der Aufbau von Genfamilien ist gesteuert und gezielt erfolgt.
Was heute an Genduplikationen abläuft, sind entweder sekundäre Ereignisse (die definitionsgemäß nichts mit dem Ursprung der primären Arten zu tun haben und sich entsprechend störend auswirken können) oder es handelt sich um regulatorische Möglichkeiten zum Aufbau von Resistenzen durch Genamplifikation, wie das bei Bakterien wiederholt beobachtet worden ist. Schmidt trennt aus diesem Grunde das Thema der Gen-Amplifikation von der Zufalls-Duplikation streng ab und stellt nach Aufführung mehrerer Punkte (wie der Genzahlen für 18s-rRNA und 28s-rRNA bei verschiedenen Organismen) 1985, p. 121 u.a. fest:
Für eine programmierte Entstehung spricht auch, daß für manche Gene die Genamplifikation zeitlich begrenzt ist, zumeist im Zusammenhang mit der Spezialisierung und Differenzierung der Zellen. Auch wenn bisher im einzelnen unbekannt ist, welche Faktoren in den einzelnen Zelltypen eine unterschiedliche Genvermehrung auslösen können, läßt auch diese Genamplifikation - quasi auf Knopfdruck und Bestellung - kaum eine andere Deutung als einen kybernetisch gesteuerten Regulationsvorgang zu. Ich kann mir jedenfalls nicht vorstellen, wie eine solche zeitlich begrenzte Genamplifikation - beim Versuch einer neodarwinistischen Interpretation - als Mutation mit Hilfe der Selektion aus einem Meer von anderen Genen hätte herausgefischt werden sollen: Wo begann der Selektionsvorteil ausreichend groß zur Durchsetzung zu werden - bei 2, 10, 100 oder 1.000 Genkopien?
Auch hier wird dem Zufall offenbar nur Raum gelassen, wo eine Steuerung keinen Vorteil bringen würde. Der Zufall bleibt damit auch hier, was er immer war. Eine Herausforderung an lebende kybernetische Systeme, aus dem gesteckten Rahmen das beste herauszuholen.
Wenn man sich erst einmal gründlich in diese Vorstellung vertieft hat, tut sich eine wahrhaft biologische Sicht der Phänomene der Evolution vor uns auf, die mit dem Lotteriespiel des Neodarwinismus nichts mehr gemein hat.
Ganz in diesem Sinne kommen Sclafani und Wechsler 1981, p. 687, bei ihren Studien der hohen Duplikationsfrequenz der dna-
b-Region von Escherichia coli zu dem Schluss: "Duplications may reflect the existance of a gene amplification control mechanism."Ergebnisse, die diese Interpretation nahelegen, haben auch Edlund et al. 1979, Winfield und Falkinghaus 1981 (p. 15: "Because of the high frequency of duplication formation and reversion in bacteria, gene duplication is well suited for providing temporary adaptation to changing conditions.") und die schon oben (p. 377) zitierten Autoren Fogel et al. 1983 u.v.a. vorgelegt.
Reversible Gen-Amplifikation könnte auch eine (zusätzliche) Heterosisursache bei Pflanzen sein (Lönnig 1980).
Amstutz et al. bemerken 1985, p. 879, zur Sequenzkonservierung bei Genfamilien:
A common feature of gene families in the eukaryotic genome is the unexpected sequence conservation between the individual genes in a given species. This cannot be explained by mutation and natural selection alone. Several rectification mechanisms have been proposed (Smith 1974).
Zu dem für ihre Beobachtungen vorgeschlagenen Korrekturmechanismus der INTERGENIC CONVERSION schreiben die Autoren:
It describes an information transfer between nonallelic genes of closely related sequence by a mechanism analogous to that proposed for recombination of allelic genes (for review, see Szostak et al., 1983). In contrast to unequal crossing-over between tandemly repeated genes, intergenic conversion does not lead to deletions and duplications. It may act on tandemly repeated sequences as well as on dispersed repeated genes.
Nach Szostak et al. 1983, p. 25, ist Genkonversion der "nicht-reziproke Informationstransfer von einer DNA-Duplex zu einer anderen in der Meiose und ist häufig mit Crossing-over verbunden" (wohlgemerkt: nicht unequal Crossing-over).
Da zum Beispiel die tRNA- und die rRNA-Gene in hoher Anzahl im Genom der verschiedensten Organismen vorkommen (bei den Wirbeltieren werden für die Ersteren mehr als 1.000 und für die verschiedenen Klassen der Letzteren 160 bis 10.000 angegeben), müssten allein schon durch Punktmutationen solche Gene in relativ kurzer Zeit in ihren Sequenzen stark auseinanderdriften, so dass das harmonische Zusammenspiel der in hoher Anzahl benötigten tRNAs und rRNAs zunehmend gefährdet wäre. Es ist daher ein Regulationsmechanismus für Aufrechterhaltung der voll funktionsfähigen Sequenzen notwendig. Wir haben hier also eine Art Reparaturmechanismus vorzuliegen, der die weitere Funktion garantiert.
Amstutz et al. erwähnen in der schon zitierten Arbeit, dass das unter vielen anderen Beispielen auch auf die Globin-Gene zutrifft.
Anstatt die Sequenzen der Genfamilien dem Zufall zu überlassen, werden sie offensichtlich in jeder Generation aufs neue überprüft und bei Abweichungen auf die die Funktionen garantierenden Normen zurückgeführt. Nicht zufällige unequal Crossing-overs und Punktmutationen haben in Verbindung mit der Selektion die Gen-Familien erschaffen und garantieren und stabilisieren ihre Funktionsfähigkeit - diese Crossing overs und Punktmutationen destabilisieren solche Systeme vielmehr in einem solchen Ausmaß, dass die Selektion hier nicht mehr weiterhilft - sondern die gegebene Information wird auf der DNA-Ebene (und durch das Processing auch auf der m-RNA-und Protein-Ebene) durch Regulations- und Reparatursysteme aufrechterhalten.
Das führt uns auf den Gedanken, dass auch solche Regulations- und Reparatur-Systeme bis zu einem bestimmten Grade degenerieren können und (zumindest) ein Teil der Pseudogene ursprünglich bestimmte (aber für das Gesamtsystem eines Organismus unter den gegebenen Umständen weitgehend entbehrliche) Funktionen hatten (vgl. das Degenerationskapitel und Ohno 1985). Wie schon wiederholt aufgeführt, können sich nicht nur neutrale, sondern auch schwach nachteilige Gene in großen Populationen anhäufen und durchsetzen (Kimura 1968, 1983 u.v.a.).
Regulationssysteme für Genamplifikationen und Endopolyploidie (mit Polytänie z.B. bei den Riesenchromosomen im Speicheldrüsengeweben von Dipterenlarven) sind auch für die Bewältigung verschiedener Aufgaben während der Ontogenese, insbesondere für bestimmte Organfunktionen, bei den unterschiedlichsten Organismen gegeben.
Bei der Polyploidie im Pflanzenreich stehen wir seit Jahrzehnten vor dem Problem, warum wir in der Natur hochpolyploide Serien bei verschiedenen Pflanzenarten antreffen, während uns die experimentelle Induktion und Reproduktion höherer Valenzgrade größte Schwierigkeiten bereiten.
Gottschalk schreibt 1984, p. 222, über experimentell erzeugte polyploide Pflanzen (Autopolyploide) u.a.:
Bei der Mehrzahl aller pflanzlichen Arten ist mit der Valenz von 4n das Leistungsoptimum nicht nur erreicht, sondern bereits überschritten. Höhere Polyploidiestufen treten in Colchicinversuchen nur selten auf. Oftmals gehen die hochpolyploiden Pflanzen frühzeitig zugrunde.
Obwohl der Spielraum bei den Allopolyploiden etwas größer ist (in einigen Fällen wurden 10, 12 und 14n erzielt, doch sind die Pflanzen meist steril und in ihrer Entwicklung so stark beeinträchtigt, dass sie in der Natur keine Chance zum Überleben hätten), ist auch hier die Diskrepanz zu den Polyploidiegraden vieler wildwachsender Pflanzenarten unübersehbar: Bei den Letzteren kommen so erstaunliche Fälle wie Ophioglossum reticulatum mit Chromosomenzahlen zwischen 2n = 870 und 1.450 vor (Valenz von nahezu 100n), Poa litorosa zeigt Werte bis zu 265 Chromosomen (38n), eine Kalanchloe-Art hat etwa 500 Chromosomen (28n). Zahlreiche hochpolyploide Serien sind aus der Natur bekannt (vgl. Gottschalk 1976, 1978, 1984; Lewis 1980; vgl. auch vorliegende Arbeit p. 159). Die Frage nach den Ursachen der Diskrepanz ist bis heute weitgehend offen. Nilsson hat 1953 dem Thema einen eigenen Abschnitt gewidmet, in dem er mehrere Autoren für die Auffassung zitiert, dass für die hohen Polyploidiegrade in der Natur genische Ursachen wirksam waren; mit anderen Worten hatten die Arten ursprünglich ein größeres genetisches Potential zur Polyploidieerzeugung als heutzutage und auch hier dürften Regulatorsysteme mit der Zeit degeneriert sein. Aufgrund der Diskrepanzen zwischen natürlichen und experimentell erzeugten Polyploiden hält Nilsson auch die Ursachen für wesensverschieden. Die Beobachtung, dass es in der Regel nicht möglich ist, die hochpolyploiden Varianten aus einer diploiden Spezies zu "resynthetisieren" spricht meines Erachtens dafür, dass hier tatsächlich ein größeres Regulationspotential verloren gegangen ist. Die Systeme konnten degenerieren, nachdem sich an verschiedenen Arealen unterschiedliche Ploidiegrade etabliert hatten und für ihr Überleben die ursprüngliche Flexibilität nicht mehr nötig war (vgl. das Prinzip auf der Rekombinationsebene pp. 125, 181-184).
Kommen wir auf die Genamplifikation bei Bakterien zurück. Zwei Punkte möchte ich zu diesem Thema noch hervorheben.
1. Die Produktion bestimmter Stoffe muss auf das Gesamtsystem abgestimmt sein. Überproduktion kann sich negativ für die betroffenen Organismen auswirken, wie das jüngst Ward und Lutgenhaus in ihrer Arbeit OVERPRODUCTION OF FtsZ INDUCES MINICELL FORMATION IN E. COLI gezeigt haben. Sie schreiben in ihrer Zusammenfassung 1985, p. 941, unter anderem:
The FtsZ gene in E. coli K-12 is an essential cell division gene. We report that a two to sevenfold increase in the level of the FtsZ protein resulted in induction of the minicell phenotype. An increase in the level of FtsZ beyond this range resulted in an inhibition of all cell division. Unlike the classical minicell mutant, the formation of minicells induced by increased levels of FtsZ did not occur at the expense of normal divisions, indicating that increasing FtsZ resulted in additional division events per cell cycle. In addition, increased FtsZ caused cell division to be initiated earlier in the cell cycle.
Vielleicht gibt es noch eine Umwelt, in der die kleineren Zellen einen Selektionsvorteil gegenüber den normal großen haben und sich eine solche Mutante durchsetzen könnte. Wie und wo aber soll sich eine Mutante mit einem "level of FtsZ beyond this range" und einer "inhibition of all cell division" durchsetzen? Wir sehen an diesem Beispiel aber wieder, dass die Möglichkeiten sich einem Limit nähern, dessen Überschreiten zum Ende der betroffenen Individuen führt.
2. Trotz der vielfach festgestellten leichten Induzierbarkeit von Genamplifikationen bei Bakterien käme wohl niemand auf den Gedanken zur Produktion ganz neuer Stoffe erst einmal - sagen wir - das
b-Galaktosidase-Gen zu duplizieren, um dann die redundante Kopie mit site-directed-mutagenesis z.B. in das Gen für Insulin umzubauen - auch wenn das prinzipiell möglich wäre (zur Wahrscheinlichkeit hingegen, mit Zufallsmutationen brauchbare Mengen der erwünschten Substanzen zu produzieren, vgl. pp. 359, 361). Dass man mit dieser Methode überdies prinzipiell keine neuen Genwirkketten aufbauen kann, haben wir mit Goldschmidt (s.o.) schon hervorgehoben.Einen weiteren Punkt möchten wir uns noch etwas näher ansehen:
Mehrere Autoren haben Gen-Amplifikationen auch in Gewebekulturen festgestellt (ein Teil der somaklonalen Variation scheint darin ihre Ursache zu finden). Ebenso sind Gen-Amplifikationen bei Pflanzenhybriden gefunden worden (Gerstel und Burns 1966, Hinweis bei Cullis 1985).
Flavell kommentiert die Frage 1985 pp. 141/142 wie folgt:
The common occurrence of tandem arrays of repeats and the major repeat differences between related species imply that large-scale amplification events are very common kinds of mutations and some are also fixed rapidly in species. Estimates of such events in mouse cells in culture have suggested that 10 % of the cells may suffer such mutations! (Bostock and Tyler-Smith, 1982; Schimpke, 1982; Hervorhebung von mir.)
Neben den auch auf chromosomaler Ebene festgestellten Amplifikationen, Deletionen und Translokationen werden für die somaklonale Variation auf der Gen-Ebene auch Transposonaktivitäten diskutiert (Cullis 1985).
Bei Protoplasten-Fusionen mit anschließender Regeneration von Individuen im Pflanzenreich kommt neben der Amplifikation und all den anderen zitierten Möglichkeiten noch hinzu, dass Genome und Plasmone recht unterschiedlicher Pflanzengattungen zusammengeführt und neben verschiedenen ontogenetischen Stadien auch ganze Pflanzen regeneriert werden können. Wie in der Anfangsphase der Mutationsgenetik wurden auch in diesem Forschungsbereich mit einem an Euphorie grenzenden Optimismus große Erfolge für die Pflanzenzüchtung erwartet. Burgess stellt 1984 in seinem Artikel THE REVOLUTION THAT FAILED nach Hinweis auf die durch Protoplastenfusionen überwundenen natürlichen Inkompatibilitätsbarrieren u. a. fest:
(p. 26:) So what happened in the intervening decade?
It must be clear to anyone remotely interested in agriculture that revolutionary new crops have not emerged.
… It would be difficult nowadays to find plant biologists ready to commit themselves to the open-ended optimism that was common ten years ago.
(p. 28:) … when very disparate parents are used as the source of protoplasts, all sorts of complication begin to arise. The two nuclei may not divide together, or one of them may not divide at all.
Und nach Aufführung verschiedener Unverträglichkeiten von Kerngenen und Organellen sowie der häufig beobachteten Chromosomenelimination, schreibt der Autor:
(p. 28:) If plants are recovered at all, they are frequently abnormal and cannot be incorporated into conventional breeding programmes. The tomato-potato hybrids is a case in point. This plant has been produced, but so far from being a useful and vigorous being bearing tomatoes and potatoes, all specimen so far examined have been slow growing and infertile. (Siehe auch Nachträge p. 567)
Zur Tomoffel (siehe auch Nachträge p. 567) vgl. auch Melchers et al. 1978. Gleba und Hoffmann geben ihrer Frustration zum Thema PLANT-GENOME ENGINEERING BY PROTOPLAST FUSION 1979, p. 547 folgendermaßen Ausdruck:
Indeed, it appears that plants and plant cells were created on the assumption that they need not be ideal objects for man's investigations.
Burgess, Gleba, Hoffmann und andere Autoren wollen damit nicht alle Hoffnungen der Protoplastenfusion für die Pflanzenzüchtung begraben, sondern eher auf die realistischen Möglichkeiten zurückweisen, die Burgess p. 28 wie folgt beschreibt:
Of course, protoplast fusion is not a useless technique. But it is being realised that its importance may be just one small part of a breeding programme.
Das gleiche gilt für die durch somaklonale Variation erhaltenen Linien. Insbesondere für Resistenzprogramme hatte man mit ungewöhnlich guten Ergebnissen gerechnet, da ja in Zellkulturen in kürzester Zeit auf engstem Raum Milliarden von Zellen auf Resistenzen getested werden können. Trotz einiger interessanter Befunde, stehen auch die bisherigen Ergebnisse zum Resistenzthema in keinem Verhältnis zu den anfänglichen Erwartungen (vgl. z.B. die Untersuchungen und entsprechenden Hinweise in Van Vloten-Doting et al., Hrsg., 1985, Foroughi-Wehr und Stolle 1985, McCoy 1987, Wakasa und Widholm 1987). Hätten die Genduplikationen den ihnen allgemein zugeschriebenen positiven evolutionären Charakter, dann wäre wohl aus den Regenerationsversuchen mit Zellkulturen allein schon auf Grund der zahlreich aufgetretenen Genduplikationentatsächlich eine Revolution in der Pflanzenzüchtung hervorgegangen.
Die oben zitierten Worte von Burgess erinnern stark an die Kommentare Leibenguths und Simmonds zur Bedeutung der Mutationszüchtung, die heute weniger als selbständige Zuchtmethode betrachtet, als vielmehr zur Ergänzung traditioneller Methoden herangezogen wird. "Nowadays we see mutation-induction simply as one technique which is occasionally useful in enlarging the genetic base of a programme in a limited and highly specific fashion." (Vgl. p. 351)
Keine dieser Methoden hat zu fertilen neuen Pflanzenspezies geführt. Keine der durch Zellfusion entstandenen "Monsterpflanzen" (Gleba und Hoffmann) hätten auch nur die geringste Chance, sich in der Natur zu behaupten. Zwar sind auch zum Thema der Protoplastenfusion anfangs starke evolutionistische Hoffnungen geäußert worden, aber abgesehen von der Tatsache, dass solche spezifischen Verfahren wie der Einsatz von Polyäthylenglykol oder: "Recently a technique has been developed for sticking protoplasts together with radio frequency waves; fusion is accomplished by a pulse of direct current" (Burgess, p. 28) sowieso nicht in der Natur vorkommen, ist von solchen Hoffnungen heute kaum mehr die Rede. Gen-Amplifikationen, die offenbar zu den häufigeren Mutationserscheinungen gehören und die ganz besonders zahlreich auch in Zellkulturen auftreten, haben daran nichts geändert. (Zur Bedeutung der Zellkulturen für die Grundlagenforschung allgemein vgl. Meins 1983).
__________________________
NACHTRAG, ARTBEGRIFF S. 558-565 (GENDUPLIKATIONEN UND INTRONS)
Die Genduplikationshypothese hat mit dem Exon-shuffling-Postulat in letzter Zeit eine neue Gestalt angenommen (Gilbert 1978 und 1985). Doolittle kommentiert 1987, pp. 919/920:
... many groups of two or more proteins that are otherwise nonhomologous share homologous domains or other structural modules that are coded for by defined exons in the corresponding genes. These examples show directly that shuffling has been involved in gene assembly.
- Worauf Beispiele folgen. Voraussetzung dafür wäre die Genduplikation (oder zumindest Exon-Duplikation plus Intronstücke), da sonst dem 'Spender-Gen' ein oder mehrere Exons und damit seine Funktion verloren ginge(n). Die Introns ermöglichen dann das Exon-shuffling, indem sie genug Spielraum für eine störungsfreie Rekombination der Exons durch ungleiches Crossing-over bereitstellen. Allerdings müsste sich das Exon-shuffling auf das redundante Duplikat beschränken.
Dillon bemerkt dazu 1987, p. 776:
Among the widely circulated hypotheses is that the intron separates the several functional or structural domains of the encoded product, and in some cases correlation between function and intron can be perceived (Blake, 1978; Gilbert, 1978). But those relationships are occasional only and provide no firm basis for drawing general conclusions. To cite one example of contradictory evidence, the lactate dehydrogenase A cistron has six (or seven) introns dividing it into seven (or eight) exons, whereas the encoded product has only three functional domains, none of the boundaries of which coincide with those of the intervening sequences(Li et al., 1985; Schriftbild von mir).
Lewin stellt 1987, p. 395 zu dieser Problematik fest:
The equation of at least some exons with protein domains supports the idea that this has been a fundamental relationship in the evolution of genes.
… many relationships between exons and protein domains do not conform to a simple equation, but they could be accounted for if events such as exon fusions have modified the ancestral structure during the evolution of nuclear genes.
Die Hypothese bedarf bereits der Hilfshypothese. Hinzu kommt, dass sie bislang die sonstige Funktionslosigkeit der Introns voraussetzt (Selfish-Gene-Hypothese für die Introns). Inzwischen sind jedoch folgende Funktionen entdeckt worden:
1. Zunahme der Genexpression bei Anwesenheit von Introns (Callis et al. 1987)
2. Bildung multipler Proteinprodukte durch alternatives Splicing (Schwarz et al. 1988)
3. Gene in Introns ("nested genes") (Hemikoff und Eghtedarzadeh 1987).
Huang hat 1986 zur Frage nach der möglichen Bedeutung der Introns folgende Themen diskutiert: (a) Introns share common splicing signals. (b) Introns may function as coding sequences. (c) Introns contain identifier sequences for tissue-specific gene expression. Ebenso zeichnen sich bestimmte Aufgaben für repetitive Sequenzen ab. Die Alu-Sequenzen beispielsweise könnten als Initiation Sites der Replikons dienen, wofür bereits experimentelle Hinweise vorliegen (Darnell et al. 1986, p. 548). Vogel und Motulsky schreiben 1986, p. 105 über die Alu-Sequenzen:
Since they have been conserved during mammalian evolution, as evidenced by their partial homology between primates (including man) and rodents, they might have an important function. (Siehe auch Munn und Mues 1988). By analogy with similar elements in other eukaryotes, such as Zea mays and Drosophila, they may be involved in gene expression, mutation (germ line and somatic), or recombination in germ and somatic cells.
Jedenfalls sind sowohl für Introns als auch für repetitive Sequenzen weit über evolutionistische Thesen hinaus eindeutige Funktionen im lebenden Gewebe gefunden worden. Durch solche Funktionen werden die evolutionistischen Intron-Deutungen zunehmend eingeschränkt, denn diese Hypothesen setzten ja voraus, dass die Introns keine weiteren konkreten Funktionen aufzuweisen hätten. Sind die Introns aber an den oben aufgeführten Funktionen maßgeblich beteiligt, dann betreffen ungleiche Crossing-overs nicht nur die Funktionen von Exons, sondern müssten auch durch Funktionsstörungen von Introns gegenselektioniert werden. Das gleiche gilt für repetitive Sequenzen. Wie im Kapitel über die Degeneration im Organismenreich gezeigt wurde (vgl. p. 403 ff.), soll das nicht heißen, dass nun unbedingt und absolut jeder vorhandenen Sequenz eine absolut lebensnotwendige Funktion zugeordnet werden muss; denn wären die Organismen nicht stark genug "gepuffert" - und das auf allen Ebenen - , dann würde jede x-beliebige kleine Störung, DNA-Verlust (vgl. Zitat Kornberg p. 323), Inversion etc. zum Exitus des betroffenen Organismus führen. Das dem nicht so ist, habe ich an den verschiedensten Stellen dieser Arbeit immer wieder deutlich gemacht.
Zu der Exon-shuffling-Hypothese ist ein weiterer wichtiger Punkt zu berücksichtigen. Die Tatsache, dass bei Bakterien die kodierenden Sequenzen eines Gens normalerweise nicht durch Introns unterbrochen werden, die homologen Gene bei Eukaryonten jedoch in der Regel Introns aufzuweisen haben, brachte die Hypothese in eine ziemlich starke Verlegenheit: Wenn die Gene durch Exon-shuffling entstanden sind, warum fehlen dann die Introns bei den Bakterien? Sind die Introns aber erst bei den Eukaryonten entstanden, dann beweist das, dass alle diese Gene nicht durch Exon-shuffling entstanden sein können; denn diese Gene waren dann ja schon vorhanden bevor es Exon-shuffling überhaupt gab! ("About 93 % of the enzyme activities listed in the International Enzyme Commission tables are known to be present in both prokaryotes and mammals" (vgl. p. 234;/Nachtrag 2001: Der Wert dürfte sich im Zuge der Neuentdeckungen der letzten Jahre zu Gunsten der Mammalia verändert haben).
Spätestens mit dem Nachweis von Intronsequenzen für gewebespezifische Genexpression ist die Exon-shuffling-Hypothese eigentlich erledigt. Nun kann man dennoch eine so schöne evolutionistische Erklärung nicht so einfach ad acta legen. Was macht man? Doolittle 1987, p. 920:
Pyruvate kinase has well-defined structural and functional domains that are clearly homologous in chicken, yeast and bacteria. Only the chicken gene has introns, but these in general define exons that encode discrete secondary structural elements of the protein (Lonberg and Gilbert 1985). Most likely this gene was assembled from exons before the eukaryote-prokaryote divergence, and introns have been lost in yeast and bacteria.
Das wiederum bedeutet, dass die Bakterien praktisch alle Introns verloren haben und das ganze Splicing-System dazu ("... introns are relicts of precellular genome-assembly processes and have since been eliminated from prokaryotes" Doolittle p. 926).
Wozu sollten aber präzellulare Genomaufbauprozesse Intronsequenzen für eukaryontisch-gewebespezifische Genexpression produzieren?
Durch ungenaues Intron-processing kann es bei Eukaryonten zu lebensgefährlichen Funktionsdefekten kommen (vgl. z.B. Gonzales et al. 1988).
Es stellt sich das Problem: Warum haben nur die Prokaryonten und manche Hefen ihre überflüssigen Introns samt und sonders verloren, nicht aber das Gros der Eukaryonten? Auf diese (gefährlichen!) Relikte hätten doch auch die Eukaryonten längst verzichten können! Wie schrieb doch F. Jacob 1973? "The genetic message, the programme of the present-day organism … resembles a text without an author, that a proof-reader has been correcting for more than two billion years, continually improving, refining and completing it, gradually eliminating all imperfections." Und da sollen die Eukaryonten seit mindestens 1,5 Milliarden Jahren Zehn- bis Hunderttausende völlig sinnlose Relikte durch ihr Genom schleppen?
Wie häufig soll das Exon-shuffling sein? Doolittle 1987, p. 922:
Although it is impossible to measure the rate of shuffling, the relative stability of gene structure (e. g., for the glycolytic enzymes just discussed) across the broadest possible phylogenetic spectrum suggests that it has been relatively infrequent since the prokaryote-eukaryote divergence.
- Womit eventuelle evolutionäre Funktionen der Introns auch noch weitgehend entfallen. Für die Beibehaltung der Introns zwecks zukünftigen Exon-shufflings müsste die Evolution jedoch über vorausschauende Fähigkeiten verfügen - was sie nach Meinung aller mir bekannten Evolutionsforscher nicht hat und auch prinzipiell nicht haben kann (vgl. Dokumentation p.570 ff.)
Man kann also die Elemente dieses Puzzles drehen und wenden wie man will, - beim Versuch sie zu einem Evolutionsbild zusammenzufügen, bleiben immer die besten Stücke draußen. Lassen sich jedoch die oben zitierten Intronfunktionen verallgemeinern, dann entfallen damit auch die bisherigen evolutionistischen Intron-Spekulationen.
Wie aber sonst ist dieses Puzzle zu erklären? Die Beschreibung der Genstrukturen ist ja korrekt, die Domänen der Proteine wiederholt beschrieben und der Aufbau der Systeme durch ähnliche Bauteile immer wieder festgestellt. Darüber hinaus findet man auch innerhalb heutiger kodierender Sequenzen Wiederholungen von oligomeren Einheiten, die Ohno (1984, 1987) zur These der EVOLUTION FROM PRIMORDIAL OLIGOMERIC REPEATS TO MODERN CODING SEQUENCES (so der Titel einer Arbeit von 1987) geführt haben. Hier als Beispiel die kodierende Sequenz des Forellen-Histons H1:

Abb. 2 aus Ohno 1987 nach Mezquita et al.
Ohno schreibt zu dieser Abbildung (p. 329).
… it will be noted that this 630-base-long coding segment contains 25 exact copies of the base pentamer CCAAG (underlined by thick solid bars), 20 % of the entire coding sequence. Furthermore, there are local concentrations of its single base deviants; e.g., one such TCAAG underscored by a thin solid bar recurs five times between 35th and l00th codons (third to fifth rows of Fig. 2). Thus, there remains little doubt that the ultimate ancestor of the H1 histone-coding sequence was the pentameric repeat shown in Fig. 1.

Abb. 1 aus Ohno 1987
Ohno stellt sich das so vor, dass sich zwei gleiche Pentamere zu einem Decamer zusammensetzten und dann durch ungleiche Paarung die nächste Replikationsrunde einleiteten: "... first unit pairing with second, and a paired segment serving as primer. As a consequence of this unequal pairing, decameric templates managed to become pentadecameric templates only after one round of replication, and this elongation process had no inherent limit" (Ohno 1987, p. 325).
Für weitere Details vergleiche man die Originalarbeiten. Wir können zusammenfassend feststellen, dass wir damit die Duplikationshypothese auf drei Ebenen vorzuliegen haben: 1. Oligomere innerhalb der Exons. 2. Die Exons als ganze. 3. Die gesamten Sequenzen von Genen.
Alle diese Theorien gehen von ganzen Serien unbewiesener Voraussetzungen aus, die hier im einzelnen zu besprechen ein weiteres umfangreiches Kapitel für sich erforderlich machen würden. (Zum Punkt 3 vgl. pp. 420 - 437) Es sei an dieser Stelle nur hervorgehoben, dass weder für das Exon-shuffling noch für Ohnos Oligomerhypothese in der hypothetischen Ursuppe experimentelle Beweise vorliegen. Zu den neuen Vorstellungen Ohnos bemerkt Salthe 1987, p. 214: "It seems to me the problem of accidental chain termination continues to be as much of a problem with this model as was with gene duplication." Zur systematisch-eindeutigen Widerlegung der Ursuppenpostulate vgl. z.B. Vollmert 1985 und Hans Krause (a) (2000): Die Zelle - Ihre Ursache, und (b) (1999): Das Leben auf der Erde - Die erste Zelle: wie entstanden? Die Voraussetzungen für Ohnos Hypothesen - base oligomer formation, and its elongation after each replication in prebiotic world - haben niemals auf unserer Erde existiert!
Es sei an dieser Stelle nur noch angemerkt, dass Prokaryonten kein Histon H1 aufzuweisen haben (die allermeisten haben überhaupt keine Histone) und dass die starke Konservierung der Histonsequenzen allgemein für spezifische eukaryontische Funktionen spricht, die in der hypothetischen Ursuppe nicht gegeben waren.
Alternative: Nehmen wir zum Beispiel folgenden Text (aus Kornberg 1980, p. 284):
Replication
Gene 32 protein is required throughout replication. It does not have the catalytic powers of the replication enzymes induced by phage infection, but is needed in amounts stoichiometric with the nascent DNA. Unlike an enzyme, the binding protein is a reactant that changes both the rate and equilibrium of the reaction. Thus the protein has a structural function, though it is not incorporated into the mature phage.
Jetzt stelle man sich vor, dass ein Analphabet, der nie etwas vom Lesen und Schreiben gehört hat, eine Hypothese zum Ursprung dieser Buchstabensequenz ausarbeiten soll (wir nehmen dabei an, dass es sich um einen Menschen mit hohem IQ handelt, der bisher nur nicht die Möglichkeit hatte, von solchen Kenntnissen zu erfahren und sich entsprechend Lese- und Schreibfertigkeiten anzueignen). Zunächst würde er feststellen, dass sich bestimmte Elemente der Sequenz laufend wiederholen, so zum Beispiel:
the: 8 mal
is: 4 mal
a/an: 3 mal
replication: 3 mal
protein: 3 mal
enzymes: 2 mal
in (induced, infection, 3 x protein, binding, incorporated): 8 mal.
etc... In Unkenntnis des wahren Sachverhalts würde er wahrscheinlich auf die Idee kommen, dass ein beträchtlicher Anteil der Sequenz durch Duplikationen von primordialen Baueinheiten und deren Derivate entstanden sei (wie z.B. das Undecamer replication und seiner Derivate replicate, replica, replications, replace, replant, replay, replenish, replenishment, replete etc. in anderen Texten, was zu solch netten Ableitungen wie punch, punk, punt, pundit von pun oder puny führen könnte!)
Ich werde einfach den Eindruck nicht los, dass wir in Unkenntnis der Grammatik und Bedeutung der Urtexte clevere Theorien erfinden, die mit der Realität nur wenig zu tun haben.
In diesem Zusammenhang sei auf einen Kommentar S. Brenners in seinem Beitrag THE DECIPHERMENT OF LINEAR G (Nature 329, 496-497; 1987) hingewiesen. Er vergleicht hier die Entschlüsselung der DNA-Sequenzen mit der der Hieroglyphen ("Linear B" von Ventris) und bemerkt: "I have always wondered whether there is more than just a joking resemblance between deciphering an ancient language and reading the genetic script.
We can view Linear G, the language of DNA, as a very ancient script - the oldest there is in fact - and some believe that there are clues to its meaning that derives from its origin and its evolution."
Dass meiner Auffassung nach die Konstruktionen der Lebensformen ihren Ursprung in intelligenter Planung, Geist und Weisheit finden, habe ich auf den Seiten 282- 290 ausführlich begründet.
Mit diesen Ausführungen soll nun nicht einer total statischen Auffassung des Genoms das Wort geredet sein. Borst und Greaves bemerken in ihrem Beitrag PROGRAMMED GENE REARRANGEMENTS ALTERING GENE EXPRESSION 1987, p. 658, unter anderem:
Such DNA rearrangements are of two types, programmed and incidental (also called unprogrammed and mutational). The incidental rearrangements arise from errors in DNA replication, repair, or recombination; from the movement of mobile elements, such as transposons; or from the insertion or excision of plasmid DNA, viral DNA, or other immigrant DNA. The exact outcome of these incidental rearrangements is impredictable, but most of them are deleterious to the individual affected by them.
In contrast, programmed rearrangements are part of the normal developmental program of an organism and its progeny. The outcome of these rearrangements is largely predictable; the process is usually carried out by specific recombination enzymes and is developmentally regulated. Such programmed (or developmental) rearrangements can be grouped in three categories: (i) amplification or deletion of genes; (ii) assembly of genes from gene segments; and (iii) DNA rearrangements that alter gene expression.
Wozu ich hier entschiedene Bedenken anmelde, das sind die 'incidental rearrangements' als einzige und letzte Quelle aller Information und Programme der gesamtem Organismenwelt (mit Hilfe der "natural rejection", wie das G. Cairns-Smith treffend umformuliert hat:"...'natural rejection', since that may be what happens most: undirected change may usually produce variant organisms which are less fit than their progenitors, and which are therefore naturally rejected, because by being less 'fit' we mean less able to survive and multiply." A. Scott (1986): The creation of life; p. 63. Oxford. Der Autor ist der Auffassung, dass beide Prozesse in der Natur ablaufen: natural selection und natural rejection, wobei die Regel offenbar mehr das letztere ist.)
Schließlich sei noch angemerkt, dass es auf die Dauer nicht besonders überzeugend ist, wenn wir unsere Wissenslücken regelmäßig im Evolutionsthesen vollstopfen, nur um sie dann proportional mit der Wissenszunahme wieder fallenzulassen, - wofür es zahlreiche Beispiele aus allen Gebieten der Biologie gibt. Zum Intron-Thema schrieb Chambon 1981, p. 95:
Es ist unwahrscheinlich, daß die heutigen Introns regulierende oder codierende Elemente enthalten, die so unentbehrlich sind, daß die durch Mutation nicht verschwinden können.
Aber warum sind sie dann noch nach 1,5 Milliarden Jahren zu Zehn bis Hunderttausenden in jedem Eukaryontengenom vorhanden?
Als einfachste Erklärung bietet sich an, daß die Introns schlicht deshalb erhalten blieben, weil die Zellen keine Möglichkeit haben, sie exakt herauszuschneiden. Dann wären die meisten Introns, wie auch ein Großteil der übrigen funktionslosen DNA in der Zelle nur ein Überbleibsel der Evolution.
Derselbe Autor geht jedoch zuvor davon aus, dass die Prokaryonten in der Lage waren, sämtliche Introns exakt und nahtlos zu entfernen:
Als schließlich alle Introns verschwunden waren, wurde auch der Spleißapparat überflüssig. Bei anderen Systemen wurde er dagegen verfeinert, als sich aus ihnen Organismen entwickelten, für die eine große Menge nichtkodierender DNA keine nennenswerte Belastung darstellte. Für diese Organismen bot das Spleißen eine wundervolle Möglichkeit aus alten Funktionen neue zu entwickeln und damit neue Wege in der Evolution zu beschreiten.
Anstatt diese lästige und gefährliche Fracht (vgl. p. 560) ein für alle Mal zu entfernen - und dass das prinzipiell möglich ist, haben die Prokaryonten mit ihrem Intron-tabula-rasa ja schlagend demonstriert - verfeinern die Eukaryonten noch ihren Spleißapparat! Das letztere war ihnen möglich, dass erstere nicht? (Quigley, Martin und Cerff sprechen 1988, p. 2676, beim Vergleich der GAPDH-Sequenzen mehrerer Eukaryonten vom "apparent massive loss of introns": Die (Kern-) Gene für Caenorhabdites elegans- und Mais-Chloroplasten-GAPDH haben im Vergleich zum Gen beim Huhn (Gallus domesticus) 9 bzw. 10 Introns "verloren". Warum aber hat das nicht auch beim Huhn geklappt? Da jedoch alle drei Organismen ein identisches Intron konserviert haben und das kaum ein Zufall sein kann, rechnen die Autoren mit der Möglichkeit eines Gen-spezifischen Selektionsdrucks, der zwischen "more and less important introns" unterscheiden konnte. Zusätzlich diskutieren die Autoren die Möglichkeit eines Neuerwerbs von Introns für die Transit-Peptid-Region. Die simple Hypothese der Introns als funktionslose Überbleibsel der Evolution noch "before the eukaryote-prokaryote divergence" (vgl. p. 560), - Überbleibsel, die nur von den Eukaryonten nicht exakt herausgeschnitten werden konnten, ist damit auch für den vorliegenden Fall als allgemeingültige Erklärung widerlegt. (Quigley, F., W. F. Martin und R. Cerff 1988: Intron conservation across the prokaryote-eukaryote boundary: Structure of the nuclear gene for chloroplast glyceraldehyde-3-phosphate dehydrogenase from Maize. Proc. Natl. Acad. Sci. 85, 2672 -2676. ) Abschließend nennt der Chambon seine Ausführungen selbst 'jedoch noch Spekulation'; denn man könne ja nicht ausschließen, dass doch noch viele Intronfunktionen entdeckt werden. - Womit wir wieder zum Beginn dieser Anmerkungen zurückkehren können.
_________________________
WEITERE NACHTRÄGE:
(GENOMMUTATIONEN BEI PFLANZEN; Artbegriff, pp. 211-212 und 545)
Durch Genommutationen entstandene Arten werden als von Lamprecht als "sekundäre Arten" bezeichnet (z.B. 1944, 1966). Diese durch Genommutationen bedingte additive Artbildung erhöht zwar die Variationsbreite, sie gibt jedoch "keinen Aufschluß über die Evolution selbst, die zur Entstehung von "Arten primären Ursprungs", zu "primären Arten" führt" (1949, p. 3).
Nach Lamprecht haben die uns bekannten Gen-, Chromosomen- und Genommutationen (in welcher Kombination auch immer) mit dem Ursprung primärer Arten nichts zu tun. Abgesehen von unserer Diskussion dieser Fragen oben, stimmen ihm weitere Autoren in mehreren Punkten zu. So gibt beispielsweise Wagner (1983, p. 427) zu Grants Buch PLANT SPECIATION u. a. zu bedenken:
There seems to be much more current interest in all phylogenetic characters and their significance than just in breeding systems and chromosome numbers. There is more and more emphasis on character divergence and shared divergent advancements as the essence of species diversification. True divergence in terms of many characters - geographical distribution, habitat, phenology, structure, physiology, chemistry - all participate in the process of speciation as well as our recognition and interpretation of taxa. In the absence of substancial divergence over the broad biological spectrum such phenomena as polyploidy, aneuploidy, sexuality, and crossability may actually be trivial, mere "evolutionary noise", things that plants do that really don't amount to much in the big picture of speciation. Ordinary diploid, sexual, outbreeding populations shall inherit the earth; it is they that supply the fundamental divergences from the groundplans of their ancestors and that underlie plant speciation, not the polyploids, the apomicts, or the hybrids. The pillars of significant speciation are character polarities and character-state correlations as they bear upon the successful adaptations of divergent plant populations, grades and clades. (Hervorhebung von mir.)
Da Wagner jedoch nicht die Möglichkeit einer Artabgrenzung durch Hybridisation von diploiden und sexuell sich fortpflanzenden Populationen ins Auge fasst (meiotisch-plasmatische Artbarriere), würde Lamprecht ihm bei der uneingeschränkten Vernachlässigung des Themas "breeding behavior" klar widersprechen.
Bleiben wir noch einen Augenblick beim Punkt "evolutionary noise":
Stebbins diskutiert die Frage nach der Bedeutung der Polyploidie unter der Überschrift "Polyploidy and the Origin of Higher Categories" (1971, p. 199):
Cytologists have often debated the question: Does polyploidy produce only new variations on old themes, or can it be responsible for major evolutionary advances? The principal argument in favour of the latter point of view has been the existence of entire tribes, subfamilies or families, (Kapitelhinweis) which have basic numbers of obvious polyploid derivation. Evidence favouring the former point of view is that most of the more advanced families of flowering plants, such as Compositae and Gramineae, contain many genera having basic numbers which are not of polyploid origin. Where genera with high basic numbers exist, they are most easily explained as the polyploid derivatives of genera having low basic numbers which already possessed the morphological specializations of the family in question. More recently, the case against polyploidy as a progressive agent has been strengthened by the increasing number of chromosome counts in woody genera of various families which are so low that they cannot be of polyploid origin. There is now good reason to believe that when chromosome numbers of the majority of tropical woody genera become known, the phylogenetic sequences of families which include genera having original diploid basic numbers will be nearly as complete as those based upon the morphology of existing forms. The existence of such sequences would indicate that polyploidy has been important in the diversification of genera and species within families, but not in the origin of the families and orders themselves.
(Zu weiteren Überlegungen des Verfassers, vergleiche man das zitierte Kapitel.)
Stebbins schreibt in ähnlicher Weise 1980, p. 165:
Polyploidie ist ein sehr häufiger Weg in der Evolution der höheren Pflanzen; zwischen einem Viertel und einem Drittel aller Blütenpflanzen sind polyploid im Verhältnis zu ihren nächsten Verwandten. Bekannte Beispiele unter den Kulturpflanzen sind Weizen, Hafer, Kartoffel, Tabak, Baumwolle, Luzerne und die meisten Arten der Weidegräser. Bekannte polyploide Unkräuter und wilde Blumen sind Iris versicolor, Thalictrum ssp., einige Wildrosenarten, Stellaria media, Montia perfoliata, Achilla ssp. und verschiedene Arten von Veilchen und Astern. Darüber hinaus gibt es Hinweise, daß vor langer Zeit Gattungen und Gruppen von Gattungen, wie Apfel, Olive, Weide, Pappel und viele Farne durch Polyploidie entstanden sind.
Trotz allem hat Polyploidie wenig zum Fortschritt der Evolution beigetragen. In Gattungen, in denen Diploide neben Polyploiden vorkommen, werden alle Haupttrends der Evolution durch diploide Arten vertreten. Die Polyploiden dienen nur dazu, die Variationen gewisser besonderer adaptiver "Themen" zu vermehren. Der Grund hierfür liegt wahrscheinlich darin, daß die große Menge der Genverdopplungen die Wirkungen der neuen Mutationen und Genkombinationen in solchem Maße verdünnen, daß die Polyploiden große Schwierigkeiten haben, tatsächlich neue adaptive Genkomplexe zu entwickeln. (Hervorhebung in beiden obigen Zitaten - bold - von mir.)
An dieser Stelle sei weiter an das oben erwähnte Phänomen der Nicht-Reproduzierbarkeit der meisten Polyploiden erinnert, das auf ein ursprünglich umfangreicheres genetisches Potential hinweist (vgl. auch die Diskussion zur grundlegenden Bedeutung dieser Frage im Gesetz der rekurrenten Variation: Cichlidae). - Beim Thema "Genmutationen" stimmen den Lamprechtschen Ausführungen ebenfalls viele Autoren zu. (Z.B. Nagel 1980, pp. 170/171; Nes und Nes 1980, p. 194; Gitt (Hrsg.) 1982; Kahle 1984 u.v.a.).
_________________________
NACHTRAG ZU SEITE 336, ARTBEGRIFF (AUFBAU VÖLLIG NEUER ENZYME DURCH GENMUTATIONEN)
Der Punkt trifft nach wie vor zu: Mir ist kein Fall bekannt, in dem Genmutationen zum Aufbau eines völlig neuen Enzyms geführt hätten. Es gibt jedoch viele Beispiele der Abwandlung von Enzymen durch Aminosäure-Substitutionen, wie ich in der vorliegenden Arbeit wiederholt betont habe. Diese Beispiele fallen unter das Gesetz der Rekurrenten Variation, - wie auch das folgende seltenere von Ohno 1984 in seinem Beitrag BIRTH OF A NEW ENZYME FROM AN ALTERNATIVE READING FRAME OF THE PREEXISTED, INTERNALLY REPETITIOUS CODING SEQUENCE (Proc. Natl. Acad. Sci. 81, 2421 - 2425) beschriebene.
Ohno weist zunächst darauf hin, dass Gen-Duplikationen für die Entstehung völlig neuer Proteinfunktionen nicht ausreichen (p. 2421):
The mechanism of gene duplication...is inherently self limiting in that a new protein arising by this mechanism invariably retains substantial amino acid sequence homology and, therefore, functional relatedness with the immediate ancestor.
Wie Ohno auch in der Zusammenfassung betont, handelt es sich bei durch Duplikationen entstandenen Genen und Proteinen um "a mere variation of the preexisted theme" (vgl. die Einwände Goldschmidts und Schmidts p. 420 ff.).
In seinem neuen Ansatz nun findet Ohno, dass durch eine Punktmutation (einer T-Insertion) ein bisheriger Leserahmen zerstört wird, aber gleichzeitig ein neuer (kürzerer) auftritt ("... the newly emerged ATG (Startcodon) created a new coding sequence from an alternative open reading frame ..." p. 2423). Funktion des "neuen" Enzyms: Abbau von "6-aminohexanoic acid cyclic dimer", einem Abfallprodukt der Nylon-Synthese (Details vgl. die Originalarbeit).
Erstens möchte ich darauf hinweisen, dass das Enzym nicht neu ist: Es bedarf keines großen mathematischen Aufwands, um festzustellen, dass diese T-Insertion im Laufe der Erdgeschichte schon Millionen von Male aufgetreten und in jeder größeren Population bereits vorhanden ist. (Vgl. Mutationstyp und Mutationsraten pp. 333, 340, 358, 372.) Erst bei reichlichem Angebot eines passenden Substrats konnten sich die Flavobakterien mit dieser Enzymfunktion in größerem Maßstab durchsetzen.
Zweitens ist die DNA-Sequenz als Alternativsequenz bereits mit der Erstsequenz gegeben, entsteht als nicht neu. Es ist weiter zu untersuchen, ob nicht allein die Tatsache, dass ein solcher alternativer Leserahmen zur Kodierung eines weiteren Enzyms überhaupt möglich ist, bereits auf eine intelligente Ursache hinweist.
Drittens ist nicht auszuschließen, dass das Alternativenzym doch noch eine Aufgabe in der natürlichen Umwelt hat oder gehabt hat.
________________________ NACHTRÄGE ZU DEN SEITEN 436/437
(PROTOPLASTENFUSION) J.E. Bravo und
D.A. Evans (1985): PROTOPLAST FUSION FOR CROP IMPROVEMENT: In: Plant
Breeding Reviews 3, 193 - 218. (Ed.: J. Janick) Westport
Connecticut. P. 202: Following the first successful regeneration of
a somatic hybrid plant, protoplast fusion was highly publicized as being
the answer to plant breeders' problems in overcoming sexual
incompatibilities. ...However, in practice, progress in the application of
protoplast fusion has been much slower than was originally expected. To
date, no new crop cultivar has been produced from breeding lines developed
using products of protoplast fusion. (Für die Zukunft sind die Autoren jedoch
optimistisch.) E. Sundberg, M. Landgren und K. Glimelius
(1987): FERTILITY AND CHROMOSOME STABILITY IN BRASSICA NAPUS
RESYNTHESISED BY PROTOPLAST FUSION. Theor. Appl. Genet. 75, 96 -
104. Nach Hinweisen auf den 'großen potentiellen Wert'
der Protoplastenfusion für den Züchter p. 96: The practical importance of somatic hybridization needs
to be evaluated. Even if an interspecific hybridization, somatic or
sexual, is successful, the utilization of the obtained hybrids can be
problematic. - Was anhand der eigenen Erfahrungen mit Brassica
napus im Detail diskutiert wird. Ihre resynthetisierten Pflanzen
zeigten im Vergleich zu den bisherigen Linien "a pollen viability of 52% -
93% and a fertility of 1% - 40%" wurde für die somatischen Hybriden
mit normaler Chromosomenzahl gefunden. Mir erschiene es realistischer, wenn man von der
"offenen Frage nach dem potentiellen Wert der Protoplastenfusion für
den Züchter" sprechen würde. Schließt man induktiv
von den bisherigen realen Ergebnissen auf die noch ausstehenden
Möglichkeiten, dann fallen die Erwartungen etwas bescheidener
aus. Y.Y. Gleba und K.M. Sytnik (1984): PROTOPLAST
FUSION. Berlin (Springer) Zur Tomoffel (p. 157): It is not out of place to recall here the optimistic
expectations of scientists hoping to produce economically valuable
hybrids by protoplast fusion. Cocking has fantasized and drawn a picture
of a hypothetical hybrid between tomato and potato that is a plant with
branches bent under the weight of tomato fruits and with stolons bearing
tubers of impressive size (Cocking 1971). There is certainly some irony in
the fact that the first parasexual hybrids of tomato (x) potato possess
neither fruits nor tubers. Melchers has recovered dozens of tomato
(x) potato somatic hybrids, a few of which are probably alloeuploids, but
no fertile hybrid plant has been found among them (Melchers, personal
communication). Zahlreiche weitere Arbeiten ließen sich
aufführen, die immer wieder die Problematik des Themas zeigen. Die
zitierten Beispiele zeigen jedoch schon zur Genüge, dass für die
Züchtung die oben (p. 437) zitierten Worte von Burgess ihre
Richtigkeit haben. Für die Ursprungsfrage zeichnet sich im
Pflanzenreich gewisse Genzen ab, die Gleba und Sytnik in ihrer oben schon
zitierten Arbeit wie folgt umreißen (p. 124): Interfamily plant cell hybrids demonstrate chromosome
reconstruction and elimination of chromosome material of one of the
species, as observed in animal somatic cell hybrids (Ringertz and Savage
1976). It is still an open question whether morphogenesis can be induced
in cell cultures of interfamily hybrids. P. 155: In our opinion, interfamily and intertribal
hybridization can not give rise to true alloeuploid plants. P. 187: Parasexual hybridization by protoplast fusion
permits the crossing of distantly related plant species which can not be
crossed sexually. Unfortunately these hybrid cells, although viable,
regenerate abnormal hybrid plants which are sterile and incapable of
sexual reproduction. Thus, the parasexual hybrids of distantly related
plant species produced to date are useless for practical plant
breeding. ...The parasexual hybrids of distantly related species
produced to date are morphologically abnormal and it is not likely that
the near future will bring the production of fertile amphieuploid
hybrids. Die Hoffnungen gehen heute mehr in Richtung auf
asymmetrische Hybriden, bei denen "small amounts of genetic material from
one species to another" erfolgreich sein sollen. Man darf vermuten, dass
auch hier noch die Schwierigkeiten nicht voll übersehen werden, die
größeren Erfolgen in der Pflanzenzüchtung im Wege
stehen. S.G. Kushnir, L.R. Shlumukov, N.J. Pogrebnyak,
S. Berger und Y. Gleba (1987): FUNCTIONAL CYBRID PLANTS POSSESSING A
NICOTIANA GENOME AND ATROPA
PLASTOME. Mol. Gen. Genet. 209, 195 - 163. (Zum Thema Somatische Hybridation p. 159:) This artificial technology was developed in a hope that
it could help to overcome the breeding barriers inherent in sexual
processes. Until now, however, fertile recombinants, though genetically
unique, have been obtained only by fusion of protoplasts of
phylogenetically close species, i.e. species belonging to the same
genus. Intergeneric and even intertribal plant hybrids have been produced
via somatic cell fusion, but none of the hybrids described to date has
been reported as being fertile... Zu ihren eigenen Befunden bemerken sie u.a. p. 162: From a preliminary evaluation of the thylakoid membrane
polypeptides of the cybrids it can he concluded that cooperation between
the Nicotiana genome and the Atropa plastome, although
functional, is not perfect. A drastically reduced content of the 27 kDa
thylakoid polypeptide is a characteristic feature of all cybrid plants
analysed. Für die Autoren ist es schon ein besonderer
Erfolg, dass hier überhaupt noch ein funktionelles System
zustandegekommen ist. Handelt es sich doch um das Zusammenführen
genetischen Materials verschiedener Triben im Sinne der
Asymmetriehypothese. Für die Ursprungsfrage ist jedenfalls von
Bedeutung, dass spätestens bei intertribalen und interfamiliären
Protoplastenfusionen sowohl Chromosomenelimination als auch
Plasmasegregation zu verzeichnen sind. Das gilt für das Tier- und
Pflanzenreich und kann als Hinweis auf die in vielen Fällen wohl
äußersten Artgrenzen im Sinne des genetisch-plasmatischen
Artbegriffs verstanden werden. Weitere Literatur: Pijnacker, L.P., M.A. Ferwerda, K.K Puite und S. Roest
(1987): Elimination of solanum phureja nucleolar chromosomes in
S. tuberosum + S. phureja somatic
hybrids. Theor. Appl. Genet. 73, 878 - 882 Evans, D.A. (1984): Isolation and genetic analysis of
somatic hybrid plants. In: Cell fusion: Gene transfer and transformation,
259 - 273. (Eds. R.F. Beers and E.G. Bassett) New York Laune, D.A. und M.D. Bennett (1987): Wide crosses
involving maize (Zea mays). In: Annual Report. Plant Breeding Institute
Cambridge.
SCHLUSSBEMERKUNG:
Dieses Kapitel könnte
noch wesentlich
erweitert und
die Unwahrscheinlichkeit des generellen Evolutionspostulats durch
Genduplikationen an Hand vieler weiterer konkreter Beispiele vertieft
werden - (so werden z.B. allein 28 Seiten Genduplikationen im Rahmen
von duplizierten Chromosomenabschnitten von D.L.Lindsley und G.G.Zimm
in
ihrem Werk THE GENOME OF Drosophila melanogaster(1992) mit oft
nachteiligem Auswirkungen auf die betroffenen Individuen
beschrieben; von
Artbildung ist hingegen dabei nirgends die Rede; vgl. in
diesem Zusammenhang nochmals den Beitrag zum Thema
Mutationen: Das
Gesetz der rekurrenten Variation). Leider ist
mir selbst die Verwirklichung einer wesentlich erweiterten Version dieses
Kapitels
wegen vielfältiger anderer wissenschaftlicher und weiterer Aufgaben
zur Zeit nicht möglich - aber vielleicht fühlt sich ein
kritischer Denker nach dem Studium der obigen Ausführungen
angesprochen, die Widerlegung der herrschenden Auffassung unter
Einbeziehung zahlreicher weiterer Fakten, zu
vertiefen! (Interessante Ansätze liefert dazu zum Beispiel
Michael J. Behe (1996): Darwin's Black Box - The Biochemical Challenge to
Evolution, pp. 89-97; The Free Press, New York. Siehe weiter Kunze,
R., Saedler, H. und Lönnig, W.-E. (1997): Plant Transposable
Elements - pp. 425-429: Gene duplications and TEs - in: Advances of
Botanical
Research 27, pp. 331-470; Academic Press, San Diego.)
Verwandte Themen/Links:
Diskussion von Einwänden zum Antibiotikaresistenz-Thema
Gregor Mendel: Why his discoveries were ignored for 35 (72) years
Wolf-Ekkehard Lönnig: Artbegriff, Evolution und Schöpfung
Ein paar offene Fragen der Evolutionstheorie sowie theologische Einwände von Evolutionstheoretikern zum Thema Intelligent Design
Wolf-Ekkehard Lönnig: Antwort an meine Kritiker
Literatur (nur Nachtrag, zum Rest vgl. die Originalarbeit):
Borst, P. und D. R. Greaves (1987): Programmed gene rearrangements altering gene expression. Science 235, 658-667
Callis, J., M. Fromm und V. Walbot (1987): Introns increase gene expression in cultured maize cells. Genes & Development 1, 1183 -1200
Chambon, P. (1981): Gestückelte Gene: ein Informations-Mosaik. In: Erbsubstanz DNA (Spektrum der Wissenschaft: Verständliche Forschung 1985), 82-95. Heidelberg. Originalartikel in Spektrum der Wissenschaft 7/1981
Darnell, J., H. Lodish und D. Baltimore (1986): Molecular cell biology. New York
Dillon, L. S. (1987): The gene. New York
Doolittle, W. F. (1987): The origin and function of intervening sequences in DNA: A review. American Naturalist 130, 915-928
Gilbert, W. (1978): Why genes in pieces? Nature 271, 501 (1985): Genes-in-pieces revisited. Science 228, 823 824
Gonzalez, F. J., R. C. Skoda, S. Kimura, M. Umeno, U. M. Zanger, D. W. Nebert, H. V. Gelboin, J. P. Hardwick und U. A. Meyer (1988): Characterization of the common genetic defect in humans deficient in debrisoquine metabolism. Nature 331, 442-446
Hemikoff, S. und M. K. Eghtedarzadeh (1987): Conserved arrangement of nested genes at the Drosophila Gast locus. Genetics 117, 711-725
Huang, P. C. (1986): Transcriptionally regulatory sequences of phylogenetic significance. In: DNA Systematics. Vol I: Evolution, 189-221. (Ed.: S. K. Dutta) Boca Raton Florida USA
Jacob, F. (1973): Zitat nach Lewin 1987, p. 397
Kornberg, A. (1980): DNA Replication. San Francisco
Krause, H. (1986): Das Leben auf der Erde - Die erste Zelle: Wie entstanden? Stuttgart (Internet-Ausgabe 1999)
Lewin, B. (1987): Genes III. New York (1995: Genes V)
Munn, T. Z. und G. I. Mues (1988): Highly conserved repeats in heat-shock introns. Nature 332, 789 ("If the repeats that occur three times each in the rat and human introns have been in place before the divergence of these two species, then the extent of sequence similarity suggests that they have a functional role.")
Ohno, S. (1984): The birth of a unique new enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence. Proc. Natl. Acad. Sci. 81, 2421 -2425
Evolution from primordial oligomeric repeats to modern coding sequences. J. Mol. Evol. 25, 325-329
Salthe, S. N. (1987): On the trail of the unknown in biology (Buchbesprechung).
J. Hered. 78, 213-214
Schwarz, T. L., B. L. Tempel, D. M. Papazian, Y. N. Jan und L. Y. Jan (1988): Multiple potassium-channel components are produced by alternative splicing at the Shaker locus in Drosophila. Nature 331, 137-143
Vogel, F. und A. G. Motulsky (1986): Human Genetics (Sec. Edition). Heidelberg
Vollmert, B. (1985): Das Molekül und das Leben. Reinbek